もうすぐ9月だというのに、まだ暑いですねえ。
私は蚊が嫌いです。 夜中でも殺すまでは寝られません。
書いている今も、どこかに蚊がいるんですよね。 「虫よけ当番」を枕元に置かなきゃ。
ま、それは置いておいて、PacBioは1分子シーケンスを売りにしています。
基本的にPCR増幅はしません。
想像はつくと思いますが、そうするとスタート時にDNAはたくさん必要になるのです。
ちなみに今は、2本鎖のDNAだけ、からスタートできます。
スタート時に必要なDNA量ですが、サイズによっても違いますが、大体、10Kbのライブラリーで読むときは、9μgくらいは必要でしょうか?
と、これはアメリカの受託会社、Expression Analysis (EA)のドキュメントが公開している数値です。
Webで公開しているので書いても良いでしょう。
ドキュメントはここ(http://www.expressionanalysis.com/platforms/category/pacific_biosciences/)
のページの、右上の、Comprehensive PacBio RS Overview というところからダウンロードできます。
実は、この会社との出会いが、私をPacBioにのめり込ませたのですが、この会社は、アメリカでマイクロアレイやシーケンサーの実験・解析受託をしています。
昨年の6月に、PacBioのサービスを始めて、安定してビジネスを展開しているそうです。
さて、DNA必要量の話に戻りますが、9 μg という数値はかなり多いでしょう。
シェアリングして、End-repairして、Ampure Washして、…と進めていくと、ライブラリーにするころには、750 ~ 1250 ng が予想される回収量になるそうです。
(なるそうです、と書きました。いえ、私も予想回収率くらい知っているんですよ。でもPacBioのプロトコールはWeb公開されていないので、ブログで書くのはまずいからあくまで第三者のふりしてます。詳しく聞きたい方は別途)
EA社はこの場合、20 ~ 35のSMRT Cellが使用できる、と書いています。
で、データはどれくらい出てくるか、ですが、10Kbで90分Movieで読んだとき、生データで最長20Kb、クオリティフィルタリングした後で14.6Kb
平均リード長は、生データで3.7Kb、フィルタリング後で2.6Kb
フィルタリング後のリード数は44,232本、塩基数は116Mb
このデータは、比較的現実に沿った数値です。
チャンピオンデータではありませんが、いろいろなサンプルをやった経験値でしょう。
フィルタリングの条件は、リードにはそれぞれRead Qualityという精度の指標があるのですが、これが 75/100 以上であることと、クオリティの良い部分のリードの長さが50bp以下ではないこと、です。
これにひっかかるリードは除去されます。
そうして残ったリードの、平均長が 2,600 bp、Maxが14,600 bp ということなんですね。
Read Quality 75以上、リード長50bp以上、というのはデフォルトの値です。
50bpでは短すぎるな、と思うときは私はこれを上げていいと思います。
サンプル調整自体は、私もトレーニングを受けましたが、それほど難しいステップはありません。
シェアリングもCovarisの機械任せだし、精製もマグネットビーズでピペット処理だし、アダプターダイマー(アダプター同士の接着)をこれまたビーズで取り除くだけ。
基本プロトコールは順調です。
でも、裏プロトコール?みたいなトラブルシュート的なプロトコールでやったときはほとんどDNAをロスってしまいました。
ラボを離れて約7年・・・の私には、裏プロトコールは難しかった。
とは言っても、基本プロトコールで作った10Kbのライブラリーの方は、ライブラリーまでの回収率は33%で、Good!
ランの結果も、上記EA社の数値と同じくらいでした。
ところで、より少ないスタートDNA量でランできないものでしょうか。
たくさんDNAがとれないサンプルでも、長く読んでみたいという要望はあります。
この要望に応えるための方法は、・・・
公式にPacのWebSiteで公開されたらここでも書こうかと思います!
まだ書けないーっ!
2012年8月27日月曜日
2012年8月2日木曜日
Base Modification 塩基修飾 (新
気づいたら8月ですね。 7月はブログ、サボっていました。
「最近更新してませんね」って言われるようになり・・・。
全然関係ないですが、西日本では、クマゼミが鳴いていますね。
沖縄からずっと愛知県くらいまではクマゼミ、見ますね。
でも東京(少なくとも私の住んでる板橋区)では見たことありません。
子供の頃から、図鑑でしか見たことのないクマゼミを、大人になって初めて見たのは京都に出張した時でした。 3年前くらいのこと。
今でも出張先で見つけると、「ああ、捕まえたいなあ。東京に持って帰りたいなあ」って思います。
さてさて、今日はBase Modificationの話です。
塩基修飾とも言います。
Pacの機械はシーケンサーなのに、DNA修飾を検出することができる!というのが売りのひとつです。
営業的には正しいのですが、過度な期待は禁物、ということで、ここではもう少し正確に書こうと思います。
今まで、「ショートリードの憂鬱」というブログの中で書いたことがあるんですが、
「メチレーションをダイレクトに検出するシーケンサー」
この中に書かれていることをちょっとだけ修正します。 まあ、こちらのブログを書いたときは、私は別の会社にいましたので、情報があまり無かった、という言い訳も成り立ちますよね。
どこを修正するかというと、後半部分に書いた、何百回も読まなくても同じ場所を5回くらい読めばメチル化と非メチル化のDNAを区別できる、という所。 これは人工配列でできた、というレベルのことで、実際にE.coliなどの配列で区別するには、もっともっと深いカバレージが必要です。
ここからが本題!
PacBioに付属する解析専用ソフト、SMRT Analysisの最近のバージョンに、新しくBase Modification 検出機能がつきました!
って言うと格好いいんですが、正確には、Base Modが起こったかも知れない場所の検出機能がつきました! というのが正しい。
どんな種類の塩基修飾なの? どれくらいの信頼性で? というところは次バージョンに期待してください。
先のブログにも書いていますが、メチル化サンプルと非メチル化サンプルとを両方持っていて、それぞれPacBioで読んだ場合、これら2つのサンプルの塩基のIPDを比較します。
IPDというのは、Inter Pulse Duration の略で、ある塩基が読まれたあと次の塩基が読まれるまでの間の時間と同じ意味です。
メチル化された塩基がテンプレートにあると、Polymeraseがポージング(ちょっと止まる・スピードが落ちる)を起こし、塩基取り込みまでに時間がかかります。
挿絵は Flusberg et al. (2010) Nature Methods 7: 461-465
例えば6mAの場合、テンプレートがmAのとき、Tが結合するまでの時間は、テンプレートがただのAの時と比べて平均5~6倍長い。その比(IPD-Ratio)を見ることで、ここに修飾が起きていそうだな、と予測できるのです。
これも同じ論文から。 6mAの場合です。 IPD-Ratioが縦軸で、横軸は塩基の場所です。
このピークの高さと並び方をシグナチャーと呼んでいます。
必ずしもメチル化されている箇所でPolymeraseがポージングするわけではなく、メチル化箇所の周囲の塩基でもポージングされることがわかっています。
そしてそのパターンが、塩基修飾の種類によって、様々に異なることもわかってきました。
PacBioでは、色々なタイプの塩基修飾、DNAダメージのIPD-Ratioシグナチャーを集めています。
(ブログでは明らかにできませんが、プレゼンでなら紹介できますので興味あるかたはお知らせ下さい)
この情報を使えば、配列を読んだだけで、IPD-Ratioのパターンから、どんなタイプの塩基修飾が起こっているのか、予測できる、というわけです。
と言っても、現バージョン(2012年8月現在)ではそこまではできません。
では、今は何ができるか!
- 読まれた部分の配列の全IPD-Ratioの情報を使って、バックグラウンドから有意なIPD-Ratioを見つける (ここに塩基修飾が起こってたかも?と期待させる)
- IPD-Ratioのコントロールは無くても良い。コンピュータで求めたin silico コントロールを使うことが可能 (これは約12塩基の長さで、全塩基組み合わせのIPDをあらかじめシュミレートしたもので、生物種を問わず使うことができる)
- 意味ありげなIPD-Ratioの場所をリストしてくれる
- そのリストは、Viewerでも表示できるし、前後20塩基の配列とともにGFFファイル、塩基単位のCSVファイルで出力
- GFFからはモチーフを見つけたりできる
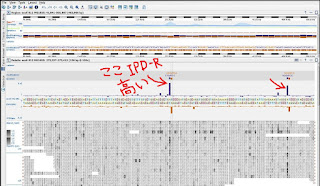
真ん中の青と赤の棒がIPD-Ratioです。 ここに塩基修飾があったかもしれない。
他の場所と比べて抜きん出ていますね。
実際このIPD‐Rが周りと比べて有意かどうかは、IPD‐Rの高さだけでなく、Coverageの深さも問題になります。
そこでCoverageについて
IPD-Rシグナチャーの種類によって大きく異なります。
はっきりしているシグナチャーを持つ塩基修飾タイプ(先の6mAなど、5-6倍のRatioを持つもの)については、15-20xが最低必要で、Pacでは50xを勧めています。
IPD-Rが2くらいの、ゆるーいシグナチャーを持つタイプは、最低200xは必要です。
ではでは、はっきりしたシグナチャーを持つ6mAを読み取ろうとした場合、どれくらいのSMRT Cellが必要なのか?
ターゲットサイズが5Mbだとします。
+/-両方のストランドを読みますから 5Mb x 2 = 10 Mb
50カバレージ必要だとすると、10Mb x 50 = 500Mb
1 SMRT Cellで100MbのMappableなシーケンスが出てくると仮定すると、500Mb / 100Mb = 5 個のSMRT Cell が必要という計算になります。
(大体1kb前後のライブラリーを作成して読むことを想定)
無論、Mappableなシーケンスの数は生物種によって様々なので、最初に1個か2個ランして確かめる必要はあります。
+/ - それぞれのストランドで50x必要、というのが忘れがちですが大事ですね。
5個だとすると、SMRT Cellは1連8個入りですから一回のランで済みます。
45分x2回のムービーでシーケンスするとして、90分 x 5 = 7.5時間
機械にセットしてからのロボットによる試薬調整、ベースコールや転送に+2時間として、ランにかかる時間は、合計10時間弱
オーバーナイトで行う感じでしょうか。
解析には2、3時間くらいかかります。
登録:
投稿 (Atom)