本当はこの話は昨年の年末に書いたのですが、ブログのサーバの不具合で投稿されていませんでしたので、再投稿。
以下のデータは12月のものです。 今はもっとたくさんありますが、それはおいおい。
さて、今の会社に入って1年が経ちました。
早いなあ、と感じる一方、1年前が3年くらい前に感じます。 ものすごく密度の濃い1年でした。
まあ、事業部の立ち上げ初年度でしたので、いろいろ手探り状態から始まり、PacBioからは毎月いや毎週かな、ひとが来る。
海外とやり取りが増えると、当たり前ですが時差があるので土曜もメールが来る。
スマホの有り難みを肌で実感です!! その分忙しくなった・・・。
海外の知り合いはとても増えました。
この業界は狭い、というのは海外も同じだということもわかりました。みんなどこかでつながっている。
他のシーケンサーメーカーもそうでしょうが、PacBioは常に進化しているので、ついていくのが大変です。
1年前に書いたことがもう時代遅れになる。
このブロブもそういうつもりで読んで頂ければ幸いです。
カタログも書き換えなくてはいけないかも。
そこで、1年前に書いたことが古い情報になる例をひとつ示します。
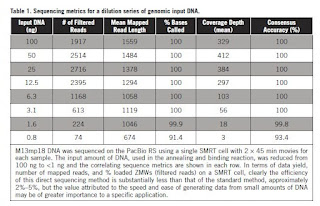
このグラフと数値は、今のカタログに出ているものです。
10kbのライブラリを、90分Movieで測定したときです。
私が昨年5月に「NGS現場の会」で喋ったとき、後から座談会みたいなテーブルで、このリードの長さは将来どれだけ伸びるか、という質問を受けました。
皆さん、ロングリードに興味のあるひとはこの点を聞いてきますね。
1.5倍くらい伸びる、と答えた記憶があります。
そしてあれから7ヶ月後、最新のデータはこちらです。 恐らくこれがオフィシャルなカタログ数値となるでしょう。
- 平均Mappedリード長: 4,500 bp
- 95th Percentile: 12,000 bp
- 最大リード長: 21,000 bp
これはPacで11kbのライブラリを、最新の試薬で120分のMovieで読んだ時のものです。
実は、XL(Extra Long) Chemistry という新しい試薬&酵素が最近リリースされました。
諸般の事情で正式リリースが遅れましたが、日本でも今随時アップグレードしつつあるところです。
そう、アップグレードによって、120分のMovieで読めるようになったのです!
われわれも実際、アップグレードして読んでみました。
11kbのコントロールを、120分で読んでいます。
上のグラフは、アダプターを取っただけの、サブリード長が横軸に、その数が左縦軸に。
まだマップさせていません。
Subread Length = 11kbのあたりにピークがありますね。
実は11kbジャストのライブラリではありません。ちょっと長いものも、短い(数千bp)ものもあります。
しかし、アダプター間の距離を見たところ、このランしたサンプルのインサートサイズで一番多いのは、11kbでした。
ではなぜ上のように、11kb未満(11000より左側)のサブリードが多数を占めているのか?
理由1: 11kbのインサートで、11kb以上読めた場合、例えば15kb読めた場合、アダプターをぐるっと回って相補鎖側の4kb(15-11= 4kb)がもうひとつのサブリードとしてカウントされる
理由2: 短いインサートは、長いMovie時間読まれれば、それだけ多くのサブリードを生産するだろう。 グラフの縦軸はサブリードの数なので、短いインサートから生産される多くのサブリードが多くを占めて見える。
からかな?
このグラフがリファレンスにマップさせた後のリード長の分布
縦軸がリードの数、横軸がMappedリード長
さて、数字は次の通り
- フィルタリング前の全ZMW数: 75,153
- そのうち解析可能なデータが出力されたZMWの割合: 28.6% (これはちょっと少なめだ)
- クオリティと長さ(50bp)でフィルタリングした後のリード数: 21,495
もうすこし多くのリード数が得られれば、もうちょっとグラフの形も変わるかも。
まあ、初回のランなのでこんなもんでしょう。
それよりリード長に注目!
- サブリード単位のマッピング精度の平均: 86.29%
- マップされたリード数: 20,193
- マップされたリードの平均長: 4,918 bp
- 95th Percentile マップされたリード長: 13,016 bp
- マップされたリードの最大リード長: 23,540 bp
確かに長い! 長くなってる
でもこれ、ラムダコントロールのサンプルですからねえ。読めて当たり前かな。
実際のサンプルで読んだらどうでしょうね。 (<-再投稿している1月現在ではバンバン読んでいます。結果はそのうちに)
Yieldはもっと改善できるでしょう。
今年は、より長くなったリードで、より多くのプロジェクトが進むといいですね。
・・・ 追記
さてさて、ここまでは「今の」スループットの話。
PacBioや私たちのプレゼンを実際に聞いたひとは、知っていると思いますが、上記に示した数値はあと数ヶ月すると変わっているでしょう。
もちろんYield(リード出力数)、リード長共に、良いほうに。
今年も忙しくなるなあ。