紅白見ながら書いてます。PPAP、ゴジラ、真田丸、ブラタモリ。
今年は面白いな。
さて今年最後の投稿は、地味なネタです。
QuiverとArrowの違いであまり知られていないこと
GitHubのサイトでもあまり詳しくは説明されていないので、少しお話しします。
【はじめに】
QuiverとArrowは、どちらも、CCS(Circular Consensus Sequence)を作るアルゴリズムに由来します。
QuiverはRSIIのデータ用、ArrowはSequelのデータ用の、コンセンサス配列作製アルゴリズムです。
SMRT Analysis v2.3 まではQuiverが標準搭載されていて、v3.x からはArrowになりました。
v3.xは、SMRT Linkというパッケージソフトになったのですが、引き続きSMRT Analysisという名前も解析用ソフトとして使われています。
SMRT LinkでもP6-C4のRSIIのデータは解析できるよう、Arrowも対応されました。
【QuiverよりArrowの方が精度が高い】
CCSを作るとき、Quiverベースのアルゴリズムでは、何回パスを重ねても平均QVフレッドスコア30くらいで精度に限界がありました。
また、ホモポリマーもうまく認識できないという欠点もあったそうです。
これは様々なクオリティ値を使ったエラーモデルが複雑すぎて余計ノイズになったとか。
そこで開発されたのがArrowというアルゴリズム。
トレーニングモデルやエラーモデルをより単純にし、またZMWごとの違いを考慮するようにした。(2016年アジアユーザグループミーティングより)
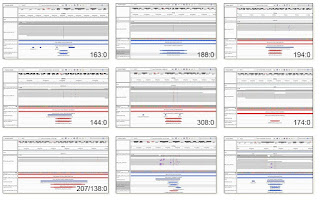
Arrowを使ったCCSは、パスを増やすとQVをフレッドスコア50、60に高めることが可能になりました。
 |
| Old = Quiver, New = Arrow |
普通、シークエンサーから出力されるリードの塩基には、その塩基をその塩基であろうとしたクオリティ値(QV)があります。
つまりAである塩基をAとコールしたとき、Cではなく、Gでもなく、Tでもなく、Aであったという「確からしさ」を表現する方法として、QVがあります。
RSIIは、ベースコールの結果、各塩基ごとのQVのほかに詳細QV (DeletionQV, InsertionQV, SubstitutionQV, SubstitutionTag, DeletionTag)という複数のQVを出力し、Quiverはこれらの情報も利用してコンセンサス配列を補正します。
Sequelは、これら詳細QVは出力せず、各塩基ごとの全体のQV(QUAL/FASTQ qv)のみを出力します。
(実データを見たことがあるかたは、現在のSequelリード BAMファイルではQVの情報が全て「!」になっているのに驚くかもしれません。この問題は次バージョンで改善される予定です)
ですがArrowではそもそも、塩基ごとのQVを使用しません!
ではArrowは何のデータをもとにエラー補正&コンセンサス作製をするのでしょう?
Arrowは、
- 各リードのシグナルノイズ比
- 各塩基のパルス幅の情報
この2つを使用して、マッピング後のコンセンサス配列を補正して作ります。
これらの情報は今もリードBAMファイルに記載が有ります。
P6-C4のRSIIデータも、Arrowを使えます。
このときは、RSIIデータ(bax.h5)をSequelのBamファイルに変換し、リードごとのSN比と各塩基のパルス幅情報を使って、Arrowが使われます。
結局、補正に使うデータを単純にしたほうが精度が上がったということでしょう。
今後、Arrowで塩基ごとのQV(詳細QVではなく、全体のQV)が使われるようになるかは、現在のところ何ともいえません。
精度が向上するようであれば、使われるようになる可能性はあります。
ああー、もうすぐ2016年も終わります。
しかし笑ったのは、白組の方がダントツで票が入ったのに何故か赤組が勝った、というアメリカ大統領選挙よりも摩訶不思議な紅白歌合戦。
明日の新聞で説明あるかな?
また来年も宜しくお願いしまーす!
2月7日の「PacBioセミナー@秋葉原」も宜しく
2月7日の「PacBioセミナー@秋葉原」も宜しく