2016年7月12日火曜日
バクテリアのIsoSeqをするには?
今日はバクテリアのIso-Seqの話です。
そもそもイントロンが無いバクテリアの転写産物ですが、完全長で読む意義はあります、よね。完全長で読みたい、というニーズはあったようです。
ではどうするか?
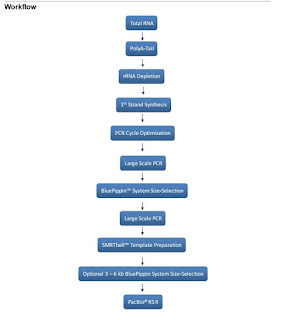
CDNAを作る逆転写には、タカラクローンテック社の SMARTer PCR cDNA Synthesis kit を使います。
でもこの逆転写反応には、PolyA-Tail 配列があることが必要です。
しかしバクテリアのmRNAには、PolyA-tail 配列が無い!
そこで最初に、mRNAの3'側に、酵素反応で強制的にPolyA-tail 配列をつけてしまいます。
Epicentre PolyA Polymerase Tailing Kit
しかしこの反応は、rRNAに対しても PolyA-Tail を付加してしまうので、この後 rRNAを除去することが必要です。
Thermo Fisher RiboMinus
流れとしてはこんな感じ
二重鎖 cDNA が得られたら、PCR増幅して、サイズセレクションして、SMRT Bell ライブラリ作って、RSIIでシークエンス
SMRT bell作る前処理の、rRNA probe とのハイブリ効率はどれくらいなんでしょう?
ここでちゃんとrRNAを除いておかないといけませんね。
プロトコルは、Unsupported つまり、サポート対象外ですが、興味のあるかたはここからご覧下さい
2016年7月11日月曜日
CCSでアセンブリも、あり!
毎日暑いですね。
そんな時はアイスかかき氷!
私は普段、冷たいものはあまり食べないですが、こうも毎日暑いと食べたくなりますよ。
「黒糖ぜんざい」
沖縄で食べたかきごおりです。ぜんざい、と呼ぶそうですが。
これはおいしかった。
さて、PacBioでのゲノムアセンブリといえば、ロングリード(CLR)を互いにエラーコレクションして行なうのが「定説」と思っている方も多いと思います。
CCSというのは、Circular Consensus Sequenceの略で、比較的短いライブラリを何度も読んで、1分子のDNAの精度を高めていく方法。
何回も同じDNAを読むことで、出力された配列のコンセンサス配列(1分子DNA由来)を、99%以上に高めることができるのです。
今の試薬P6-C4なら、5パス以上で99%の精度に達することも可能です。
ライブラリの長さにもよりますが、1kbから2kbなら、十分精度の高いリード(CCS)を出力することも可能で、それを使ってアセンブリを行なった論文がこちら
8個のSMRT Cell から得られた平均 1,319 bp、99.7%の精度(平均10パス)、94 Mb分のCCSを使って、アセンブリ。
アセンブラーは、MIRA 4.0
この論文はメタゲノムなので、完全ゲノム配列を作るのが目的ではありません。
マーカー遺伝子が読めていればOK
Phylotype Specific な配列データベースを作成するには、どんな配列もバイアス無く読めるPacBioが最適というわけです。
CCSを使った、アセンブリの論文でした。
そんな時はアイスかかき氷!
私は普段、冷たいものはあまり食べないですが、こうも毎日暑いと食べたくなりますよ。
「黒糖ぜんざい」
沖縄で食べたかきごおりです。ぜんざい、と呼ぶそうですが。
これはおいしかった。
さて、PacBioでのゲノムアセンブリといえば、ロングリード(CLR)を互いにエラーコレクションして行なうのが「定説」と思っている方も多いと思います。
CCSというのは、Circular Consensus Sequenceの略で、比較的短いライブラリを何度も読んで、1分子のDNAの精度を高めていく方法。
何回も同じDNAを読むことで、出力された配列のコンセンサス配列(1分子DNA由来)を、99%以上に高めることができるのです。
今の試薬P6-C4なら、5パス以上で99%の精度に達することも可能です。
ライブラリの長さにもよりますが、1kbから2kbなら、十分精度の高いリード(CCS)を出力することも可能で、それを使ってアセンブリを行なった論文がこちら
8個のSMRT Cell から得られた平均 1,319 bp、99.7%の精度(平均10パス)、94 Mb分のCCSを使って、アセンブリ。
アセンブラーは、MIRA 4.0
この論文はメタゲノムなので、完全ゲノム配列を作るのが目的ではありません。
マーカー遺伝子が読めていればOK
Phylotype Specific な配列データベースを作成するには、どんな配列もバイアス無く読めるPacBioが最適というわけです。
CCSを使った、アセンブリの論文でした。
2016年7月6日水曜日
パイナップルドラフトゲノム PacBioでアセンブリ
皆さん、パイナップルって、よく食べます?(私はたまーにしか食べません)
日本では、リンゴやブドウほどメジャーではないせいか、パイナップルの品種名って、あまり知らないですよね。
八百屋やスーパーでも、普通に「パイナップル」だったりするし。
恥ずかしながら、今日まで、パイナップルにたくさんの種類があるなんて気にしたこともありませんでした。
サイトはこちら
世界で一番多く栽培されているのが、Smooth Cayenne(スムース・カイエン)種だそうです。
でも今、MD-2、別名ゴールデン・パイナップル、スーパー・スイート、などと呼ばれている品種が次々と栽培されているらしい。
時は1961年、ドール社、デルモンテ社、マウイ・パイナップル・カンパニーの出資によって、ハワイ州に、Pineapple Research Institute(PRI)が設立されました。
そこで品種改良によって生まれたのが、このMD-2品種。MDの名は、パイナップル・カンパニーのゼネラルマネージャーの妻の名前、Millie Dillardさんに由来するそうです。
(以上、UTAR AGRICULTURE SCIENCE JOURNAL ● VOL. 1 NO. 4. OCTOBER 2015からの情報でした)
パイナップルって、今まで考えたことなかったのですが、受粉しなくても果実を作るそうですね。
Parthenocarpy(単為結果)と呼ぶそうです。バナナと同じ。
そして、non-climacteric な植物。
これは、果実を収穫してからエチレンガスをほとんどあるいは全く出さないので、収穫後に果実が熟すことはないらしい。ここのサイトが詳しい。
そういえば、収穫後にエチレンガスを噴霧して、食べ頃をコントロールする話は、昔聞いたことがありました
そんなパイナップルですが、MD-2のドラフトゲノムが論文になりました。
Biotechnology Research Institute, University Malaysia Sabah の、Dr.Redwan らの仕事です
PacBioを使ったアセンブリプロジェクトです。
ここではイルミナリードも使われていますが、目的はPacリードのエラー補正と、Pacアセンブリ後のContigの、Scaffoldingのみです。
先ず、イルミナライブラリ用に350bpと550bpのインサートを作り、HiSeqを使って100bpのシングルまたはペアエンドシークエンス。
さらに、750bpのインサートをMiSeqを使って300bpのヘアエンドシークエンス。
Q20のクオリティ、最低50bpのリードを解析に使用して、154.7カバレッジのデータ量を得た。
PacBioは20kbライブラリ、P4-C2、P5-C3で32セルシークエンス。
フィルタリング後の11.78Gbデータを、イルミナの350pb、750bpライブラリデータで補正。
これには、novoLR package(Novocraft ソフトウェア)を使用。
補正後のデータは8.34Gb(15.9 x)になったので、これをCelera Assemblerにかけてアセンブリ → N50 = 25,277bp
Contigの冗長性を除くため、25,000bp以下の短いContigをそれ以上の長いContigにマップして80%以上のContigを除いたり、エラー補正後のリードをContig配列にマップしたりして16%冗長配列を除いている。
このような努力の結果、トータルのContigサイズは想定ゲノムサイズの 96.6% の 508 Mb、N50 は34,762 bpになった。
このようにして作られたContigは、次にイルミナリード、補正前のPacBioリードを利用して、novoLRpolishというソフトでScaffoldingし、N50 が153,084bp、最大Scaffoldが1.29Mbになり、トータル塩基数は524Mbpになった。
アセンブル後のゲノム配列の評価には、CEGMA(Core Eukaryotic Genes Mapping Approach)というツールを使っている。
簡単に言うと、真核生物に存在するコアな遺伝子たちがちゃんと読めているか、を評価するデータツールです。
しかし、残念ながら、CEGMAは昨年サービスを中止したらしいです。
これからはBUSCO(Benchmarking Universal Single-Copy Orthologs)らしいです。
さてさて、もうひとつ今日驚いたこと。
パイナップルの果肉は、果実じゃない!
ちなみにパイナップルを使った料理のレシピは、クックパッドによれば、現在6,000種類以上もあります!!
こちらも驚き!
2016年7月1日金曜日
Iso-Seq アイソフォームシークエンスアップデート 植物
早いものでもう7月!
あー、もう今年も後半戦になっちゃったー。
7月が終われば夏休みの8月、それが終われば超忙しい学会シーズンの9月、10月。
11月になったらもう分生で、あっという間に12月で師走。
何でしょうね、歳を取ると月日が経つのが早く感じるって言いますね。
さて、アイソフォームシークエンス(Iso-Seq)で最近Nature Communicationから論文が出ました。
Iso-Seqといえば、PacBioのロングリードが活かせるアプリケーションのひとつ、完全長cDNAシークエンスです。
これは前にもいろいろ書きましたが、いわゆるAlternative Splicingを含むmRNAの構造変異が、連続配列として観測できる実験方法です。
これはとうもろこし
論文リンクはこちら
そしてこれはソルガムキビ
論文リンクはこちら
どちらも植物ですね。
とうもろこしの方は、6種類の組織から読まれた111,151種類のRNA転写産物のうち、57%はこれまで知られていなかったらしいです!!
とうもろこしといえばアメリカの主要な農作物。
もうとっくに研究し尽くされていて当然と思っていましたが、そうではなかったんですね。
ソルガムキビ、私もよく知らない植物ですが、この論文のすごいのは、彼ら独自で解析パイプラインを作ったこと。
Transcriptome Analysis Pipeline for Isoform Sequencing(TAPIS)というツールは、リファレンス配列にクオリティをパスしたCCSをGMAPでマッピングした後、リファレンス配列を使ってエラーコレクションをするらしいです。
これは新たな方法ですね。
9月24日(土)、鳥取大学にて、育種学会があります。
私はその中のセッションにて、Iso-Seqについて話す予定です。
このように新たな論文が次々に出てくるのはうれしいですね。フォローアップをちゃんとしなきゃ。
その他、Iso-Seqの論文をまとめたサイトはこちらにあります!
Pacの社員、リーズさん(かわいい台湾出身の女性インフォマティシャン)がまとめてくれました。
参考になると思います。
あー、もう今年も後半戦になっちゃったー。
7月が終われば夏休みの8月、それが終われば超忙しい学会シーズンの9月、10月。
11月になったらもう分生で、あっという間に12月で師走。
何でしょうね、歳を取ると月日が経つのが早く感じるって言いますね。
さて、アイソフォームシークエンス(Iso-Seq)で最近Nature Communicationから論文が出ました。
Iso-Seqといえば、PacBioのロングリードが活かせるアプリケーションのひとつ、完全長cDNAシークエンスです。
これは前にもいろいろ書きましたが、いわゆるAlternative Splicingを含むmRNAの構造変異が、連続配列として観測できる実験方法です。
これはとうもろこし
論文リンクはこちら
そしてこれはソルガムキビ
論文リンクはこちら
どちらも植物ですね。
とうもろこしの方は、6種類の組織から読まれた111,151種類のRNA転写産物のうち、57%はこれまで知られていなかったらしいです!!
とうもろこしといえばアメリカの主要な農作物。
もうとっくに研究し尽くされていて当然と思っていましたが、そうではなかったんですね。
ソルガムキビ、私もよく知らない植物ですが、この論文のすごいのは、彼ら独自で解析パイプラインを作ったこと。
Transcriptome Analysis Pipeline for Isoform Sequencing(TAPIS)というツールは、リファレンス配列にクオリティをパスしたCCSをGMAPでマッピングした後、リファレンス配列を使ってエラーコレクションをするらしいです。
 |
| ソルガムキビの論文より引用 |
これは新たな方法ですね。
9月24日(土)、鳥取大学にて、育種学会があります。
私はその中のセッションにて、Iso-Seqについて話す予定です。
このように新たな論文が次々に出てくるのはうれしいですね。フォローアップをちゃんとしなきゃ。
その他、Iso-Seqの論文をまとめたサイトはこちらにあります!
Pacの社員、リーズさん(かわいい台湾出身の女性インフォマティシャン)がまとめてくれました。
参考になると思います。
登録:
投稿 (Atom)