メリー・クリスマス!
いかがお過ごしですか?
こんな深夜(2時)ですがハイテンションです!
バイオインフォやってる人には「ついに!」というニュースが飛び込んできました!
もうご存知ですって!?
http://genomeref.blogspot.jp/2013/12/announcing-grch38.html
ヒトゲノム GRCh38がついにリリースされました!
前回のバージョンが2009年だからもう5年も、大きなバージョンアップはされていなかったわけです。
さてこれで、ヒトゲノム解析やっている人たちは、マッピング用のリファレンス配列を38にするのでしょうね。
何せ5年ぶりの大改訂。1000人ゲノムのデータも参照して今までのv.37からエラーと思しき配列を除いたり、ミトコンドリアゲノム、セントロメアの予測配列なども加わり、質も向上。 らしい。
アノテーションはまだありません。UCSCやNCBIからブラウザで見れるようになるにはまだあと数週間から一月くらいかかるそうです。
解析受託サービスの会社や、ソフトウェアメーカーはきっと新年早々、大忙しになるんでしょうね。
私も前職で経験あるから、わかるー。
さて、話はガラッと変わって、シークエンサーの臨床への応用。
今年はIllumina社がMiSeq Dxという機械をアメリカでリリース。FDA承認の初のNGSとして注目を浴びました。
ボストンのアメリカ人類遺伝学会で、実機を見てきましたが、見た目は普通のMiSeqと同じ。
cystic fibrosis の診断キットを同時にリリース(これがFDA認可)したことで、アメリカではいよいよNGSが臨床の現場で使われ始めたか、という盛り上がりが感じられました。
では、PacBioが診断に使われるとしたら、どんな分野が可能か?
前から注目されていたのが、トリヌクレオチドリピートの疾患です。
Fragile X シンドローム(日本語では、脆弱X症候群)という遺伝性精神疾患があり、これは、FMR1遺伝子にあるCGGリピートの数が原因です。
健常人は、6~46リピートで、200リピートになると正常なタンパク質が作れなくなり、脳の発達に異常をきたすそうです(ウィキペディアより)。
55-200リピートだとpremutationと言われ、発症はしないけれども、保因者ということになるそうです。
最近、そのCGGリピートの中に、AGGというリピートが混じることがわかって、それがあるとその子供が発症する確率が低くなることがわかったそうです。
現在は、CGGリピート配列全体を読むことは、PacBio以外のシークエンサーではできません。
AGGが含まれているのかどうかを知ることも、他のシークエンサーではできません。
そこで、PacBioをうまく診断に使えないか、という試みがUC Davisの Paul Hagerman博士のラボで行われています。
現状の課題は、CGGリピート配列をPCR増幅無しに取ってくる技術を確立すること、です。
http://www.bio-itworld.com/2013/12/18/fragile-expedition.html
2013年12月25日水曜日
2013年12月24日火曜日
年越し前に、今年最後のアップデートの話
NGS現場の会・第3回研究会で、PacBioのリードがあと2か月で平均8.5Kbpに伸びる、と発表しました。
試薬・酵素と、Movie時間のアップデートで可能になるのです。 もうリリースしてユーザのアップデートもほぼ完了したのでこの話は年明け前にしておかないと。
先ず、試薬・酵素
PacBioの酵素はPolymeraseでP、試薬はChemistryでC、と表します。
組み合わせは、P4C2とか、P5C3とか、で略します。
といっても、少し前まで、XL酵素とXL試薬、C2酵素とC2試薬という厄介な名前のものがあり、XLC2、XLXL、C2C2と略すとどっちがどっちだ!ということも良く聞かれました。
ちなみにXLC2はXL酵素とC2試薬、XLXLはXL酵素とXL試薬、C2C2はC2酵素とC2試薬の略です。
それがややこしいというのはアメリカでもクレームがあったらしく、これからはPとCではっきり分かるようにさせたということらしい。
それはともかく、今のシークエンスに使われているのは、P4C2です。
P4は、2013年の夏ごろリリースした酵素です。
C2酵素よりも安定してロングリードを作ることができる酵素です。
P4C2で読んだとき、120分Movieでは平均リード長4500bpを出していました。
C2試薬(酵素では無い)は、ヌクレオチドと蛍光色素の距離が近く、レーザーが照射したとき、蛍光色素からのエネルギーが酵素に移動するため、それによって酵素の活性が失われやすいという欠点がありました。
そこで、ヌクレオチドと蛍光色素の間に、分子を入れて、レーザーによる励起エネルギーが酵素に届かないように改善、これがC3試薬の登場です!
左側がC2試薬と酵素X、右側がC3試薬と酵素X
Protecting Scaffoldというのが、ヌクレオチドと蛍光色素の間に新たに挿入した分子です。
これがC3
C3試薬を使うと、レーザー光によってDye(蛍光色素)が出すエネルギーが、Protective Scaffold(分子)によって遮られ、酵素には届かない、という図
こうしてC3試薬が生まれ、酵素との組み合わせ実験がR&Dにて行われました。
P4酵素とC3試薬はあまり組み合わせが良くなかったのです。
夏前からいろんな酵素を試していて、最終的に残った、一番成績の良い酵素君が、P5と名付けられました。
次にMovie時間アップデートの話
今までは120分がMovie時間のMaxでしたが、これを180分まで読めるようにしました。
Movie時間を変えるのはソフトウェアをちょっと変えれば可能なので、実は、酵素が180分たっても大丈夫、になったことの方が大きい。
120分で読んだ時の1.5倍かかるわけですから、1日何セル読めるかは、新たに計算する必要がありますね。
今までは120分Movieで8セル読むとすると、機械にセットしてからデータ転送が終わるまで20時間くらいでした。180分Movieだと、おおよそ28時間。
以下のグラフは、カタログでも登場するものです。
大腸菌ゲノムで、20Kbpライブラリを作り、Blue Pippinでサイズセレクションして、180分Movieで読んだ時の平均データです。
平均リード長5500bpのP4C2は、今まで通り、デノボアセンブリやcDNAシークエンスなどに向いています。
同8500bpのP5C3は、精度がP4C2に比べて低いので、今のところスキャフォールドへの使用を推奨しています。
私たちもそのうち、こうしたデータを公開しようと思います。
でも、ポリメラーゼリード単位のデータでは、へー、で終わってしまう。
やっぱりサブリードのグラフも欲しいし、実際アセンブリした結果も欲しい。
なので、公開するときはそういうデータも一緒に。
試薬・酵素と、Movie時間のアップデートで可能になるのです。 もうリリースしてユーザのアップデートもほぼ完了したのでこの話は年明け前にしておかないと。
先ず、試薬・酵素
PacBioの酵素はPolymeraseでP、試薬はChemistryでC、と表します。
組み合わせは、P4C2とか、P5C3とか、で略します。
といっても、少し前まで、XL酵素とXL試薬、C2酵素とC2試薬という厄介な名前のものがあり、XLC2、XLXL、C2C2と略すとどっちがどっちだ!ということも良く聞かれました。
ちなみにXLC2はXL酵素とC2試薬、XLXLはXL酵素とXL試薬、C2C2はC2酵素とC2試薬の略です。
それがややこしいというのはアメリカでもクレームがあったらしく、これからはPとCではっきり分かるようにさせたということらしい。
それはともかく、今のシークエンスに使われているのは、P4C2です。
P4は、2013年の夏ごろリリースした酵素です。
C2酵素よりも安定してロングリードを作ることができる酵素です。
P4C2で読んだとき、120分Movieでは平均リード長4500bpを出していました。
C2試薬(酵素では無い)は、ヌクレオチドと蛍光色素の距離が近く、レーザーが照射したとき、蛍光色素からのエネルギーが酵素に移動するため、それによって酵素の活性が失われやすいという欠点がありました。
そこで、ヌクレオチドと蛍光色素の間に、分子を入れて、レーザーによる励起エネルギーが酵素に届かないように改善、これがC3試薬の登場です!
左側がC2試薬と酵素X、右側がC3試薬と酵素X
Protecting Scaffoldというのが、ヌクレオチドと蛍光色素の間に新たに挿入した分子です。
これがC3
C3試薬を使うと、レーザー光によってDye(蛍光色素)が出すエネルギーが、Protective Scaffold(分子)によって遮られ、酵素には届かない、という図
こうしてC3試薬が生まれ、酵素との組み合わせ実験がR&Dにて行われました。
P4酵素とC3試薬はあまり組み合わせが良くなかったのです。
夏前からいろんな酵素を試していて、最終的に残った、一番成績の良い酵素君が、P5と名付けられました。
次にMovie時間アップデートの話
今までは120分がMovie時間のMaxでしたが、これを180分まで読めるようにしました。
Movie時間を変えるのはソフトウェアをちょっと変えれば可能なので、実は、酵素が180分たっても大丈夫、になったことの方が大きい。
120分で読んだ時の1.5倍かかるわけですから、1日何セル読めるかは、新たに計算する必要がありますね。
今までは120分Movieで8セル読むとすると、機械にセットしてからデータ転送が終わるまで20時間くらいでした。180分Movieだと、おおよそ28時間。
以下のグラフは、カタログでも登場するものです。
大腸菌ゲノムで、20Kbpライブラリを作り、Blue Pippinでサイズセレクションして、180分Movieで読んだ時の平均データです。
平均リード長5500bpのP4C2は、今まで通り、デノボアセンブリやcDNAシークエンスなどに向いています。
同8500bpのP5C3は、精度がP4C2に比べて低いので、今のところスキャフォールドへの使用を推奨しています。
私たちもそのうち、こうしたデータを公開しようと思います。
でも、ポリメラーゼリード単位のデータでは、へー、で終わってしまう。
やっぱりサブリードのグラフも欲しいし、実際アセンブリした結果も欲しい。
なので、公開するときはそういうデータも一緒に。
2013年12月22日日曜日
Moleculoのリード?
丸の内KITTE(キッテ)の中にある、クリスマスツリーです!
東京駅の丸の内南口すぐ近く、日本郵政のビルにあります。
本物の雪ではないんですけど、それらしく見えますね。
私たちは2年ほど前から大手町にオフィスを構えていたのですが、このたび引っ越ししました。
会社のオフィスの引っ越しは普通でも大変だと思うのに、年の瀬の引っ越しとは・・・。
新オフィスは文京区の根津です。前ほど広くは無いですが、働くスペースとしては十分。
大手町は、それなりに楽しい街でしたが、根津もまた、下町でいい感じ。
前職の職場が人形町だったので、それに近い雰囲気がありますね。
さて、12月は2日の週は神戸で分子生物学会がありましたね。
PacBioのセミナーは行いませんでした。分生ではNGSの発表は少なかった気がします。
そんな中でも面白かったのは、Illumina社のランチョンセミナー。
Moleculoテクロノジーの話が登場しました。
東京駅の丸の内南口すぐ近く、日本郵政のビルにあります。
本物の雪ではないんですけど、それらしく見えますね。
私たちは2年ほど前から大手町にオフィスを構えていたのですが、このたび引っ越ししました。
会社のオフィスの引っ越しは普通でも大変だと思うのに、年の瀬の引っ越しとは・・・。
新オフィスは文京区の根津です。前ほど広くは無いですが、働くスペースとしては十分。
大手町は、それなりに楽しい街でしたが、根津もまた、下町でいい感じ。
前職の職場が人形町だったので、それに近い雰囲気がありますね。
さて、12月は2日の週は神戸で分子生物学会がありましたね。
PacBioのセミナーは行いませんでした。分生ではNGSの発表は少なかった気がします。
そんな中でも面白かったのは、Illumina社のランチョンセミナー。
Moleculoテクロノジーの話が登場しました。
初めて聞くひとも多かったと思います。
その中で、Moleculoのロングリードは、平均何キロbpとか、精度がQVいくつとかの数字が出てきました。
Moleculoのテクノロジーは、それ自体は素晴らしいものだと思います。 ショートリードのシークエンサーで、ロングリードを再現させる、という発想はさすがアメリカのベンチャーです。
技術の全ては明らかにされていませんが、10Kbpにゲノムを断片化したあと、ウェルごとに数百の断片化クローン配列を増幅し、クローンごとにタグ付してから、HiSeqでシークエンスします。
そのあと、タグごとにリードを分けてアセンブリし、もとのクローン配列(10Kbpに断片化した配列)を再現するというわけです。
しかし、
誤解を恐れずに言えば、Moleculoが出すデータはContigです。
「リード長10Kbpのデータを出力できます!」 というのは、正確には「元々10Kbpに断片化した配列をMoleculoテクノロジーを使ってタグ付してショートリードで読み、タグを基準にそれぞれをアセンブルすると、10Kbp長のContigが再生できます!」
ということ。
再生されるContigは、(アセンブルのデータなので当たり前ですが)全てが10Kbpというわけでは無い。
やはり短いContigがたくさん出てきて、配列が長くなるにしたがってContig数は少なくなるという普通のアセンブリの傾向があります。
というわけで、MoleculoのContigリードと、PacBioのリアルリードを、配列単位の長さや精度で比較するのはフェアでない。
と思うのです。
しかしこれ以上書くといろいろとまあアレなので、以上。
2013年11月28日木曜日
アーバスキュラー菌根ゲノムからわかったこと
私たちの細胞には、ミトコンドリアがあって、これが独自のゲノムを持っていて、エネルギーを作り出している云々。
その時素直に、「へえー、生物の中にも生物が住んでいるんだあ」と驚いた記憶があります。
それから腸の中には腸内細菌が住んでいて、やつらが何をしているのかはわからないが、定期的にヤクルト飲んでいれば健康でいられると、そのままTVコマーシャルを信じていた学生時代。
大学では、真菌(Fungi)という生き物の不思議さに大変驚きました。
核がたくさんあるやつや、HaploidとDiploidが人生の半々くらいなやつ。
酒造や発酵に必須で、古くから人類の文化と切っても切れない関係のやつ。
はたまた抗生物質を作ってくれて人類を病気から救ってくれたやつ。
しかし現代ではカビキラーによって、見つけ次第排除されるかわいそうな運命になっているやつ。
そして共生
植物の根っこに共生するアーバスキュラー菌根(Arbuscular mycorrhiza fungus)という種類があります。
その一種である、Rhizophagus irregularis のゲノムが、国際チームによって読まれました。
この生物、155MbのゲノムサイズでHaploid
ニンジンの根っこに共生した株から取り出したゲノムを、Sanger、454、Illumina、そしてわれらがPacBioで読んだそうです。
IlluminaはHiSeq2000を使用し、シングルエンド100bpを37,094,828本出力。
PacBioはXL-C2ケミストリーで、3.5μgのゲノムからライブラリを作成、SMRT Cellを9つ使用し、120分Movieでラン。
出力結果は、Max22kb、平均3kbのリードが139,080本。総塩基数 766 Mbp (デフォルトクオリティフィルタリング後の値)
少ないとお思いでしょうが、これは150Kアップグレードする前の装置で読んだ数字です。
その頃は、1セルあたりの出力が100 Mbp 弱だった。
ま、とにかく、9セル使って120分で読んで、155 Mbp ゲノムのおよそ5xはカバーできたわけです。
今なら、セル数は半分の4つくらいでこれくらい出ますかね。
アセンブリの方法は、先ず、Sanger、454、Illuminaのデータをまとめて、一度フィルターにかけます。
この真菌はATリッチなので、GCが45%以上あるリードはバクテリア由来のコンタミらしい、として、別途分けておくフィルターです。
残ったリードが約3000万本、3,781 Mbp
これを、CLC-Bio Genomics WorkBench のde novo hybrid assembly にかけ、91 Mbp、28,371本のScaffold(N50 = 4.19 kb)を得たそうです。
さらに、このScaffoldと、PacBioの5x分のロングリードを混ぜて、PacBioのAHA(A Hybrid Assembler)でさらにScaffoldingし、101 Mb、12,421本のScaffold(N50 = 15.16 kb)を得ています。
それでも12,000本、N50 が15 kbpかあ・・・
今なら、20kbライブラリも可能だし、180 Movie もあるし、新酵素もあるし、解析ではPB Jelly 2 という別のPac用Scaffoldツールもあるので、それらを試すともっとアセンブリも改善するでしょうね。
(もしかして現在進行中かも)
Tisserant et al. "The genome of an arbuscular mycorrhizal fungus provides insights into the oldest plant symbiosis" PNAS, Suppl. Fig S1 より
PacBioはゲノムアセンブリにしか使われていませんが、ショートリードのアセンブリ結果から、遺伝子配列の解析もされています。
その結果、 植物に共生する真菌らしく、、彼らの遺伝子セットからは、細胞壁を消化する代謝酵素遺伝子が失われていたり、リンの吸収に役割のある遺伝子がたくさん含まれていたり、そのようなことがわかったそうです。
2013年11月26日火曜日
ISBA 2013 一分子シークエンサーとナノポアテクノロジー
先週、DNA一分子のシークエンス、ナノポアテクノロジーに関しての国際シンポジウムが京都で行われました。
私もちょうど前日まで関西方面に来ていたので、京都、三十三間堂となりのHyattホテルの会場にお邪魔しました。
京都は何度も来ているんですが、三十三間堂側は初めて。
このシンポジウムに来た理由は、PacBioの創設者の一人、Steve Turner氏の講演があるから&私の友人も講演するからです。
Steveとは半年ぶりに会いました。
彼の話は、ZMWの考案にいたる歴史、つまり最初はDNAのシークエンスではなく、ポリメラーゼの動きについて研究していたところ、「副産物」として、シークエンサーのアイデアが思いついた、という話、から始まりました。
彼の専門は物理なので、ZMWについては思い入れがあるのでしょう。
それから1分子シークエンサーとして、機械を完成させ、コマーシャルラインに乗せ、超ロングリードというユニークな分野で優位を保っていること。
さらにロングリードを使ったアプリケーションを紹介していました。
彼のプレゼンは、内容もさることながら、その発表のスタイルが上手です。
喋り方がゆっくりで、発音もきれい。 ジェスチャーも自然で無駄な動きが無い。
TEDのプレゼンターもそうですが、間の取り方も上手です。
Steveは、私が見習いたいプレゼンターの一人です。
さて、ほかの参加者の話は、というと、生物系から情報処理系、完全なナノ物理系、とテーマが幅広かったです。
正直、ナノ物理系の話は30%くらいしか理解できませんでしたが、DNA分子を穴(ポア)に入れて、その分子が通る速度をコントロールすることがとても難しいということはわかりました。
穴の直径、素材、コーティング材、電圧、その他もろもろ、研究者のみなさんは相当苦労されているんですね。
単純にポアに通して・・・という話にはならず、通っても検出限界を超えるスピードで通り過ぎてしまっては意味が無い。
全ての分子が、等速度で、検出範囲内のスピードでポアを通過して欲しい。
今はまだR&Dのレベルですが、いずれマーケットに出てくる時代がくるかもしれません。
その時はどんなナノポアテクノロジーが残っているのか、興味があります。
2013年11月15日金曜日
PacBio スライドショー ASHGとUGM、最近のCSHL
人間の体温は普段と1,2度変わっただけで、体調がすごく悪くなるんですね。
先日、熱出しました。(前回はシンガポール帰国直後)
PacBioのシークエンサーも、内部の温度を常に監視していて、ちょっと異常があると警告メッセージを出すんですが、人間の体も具合が悪くなる前に軽い警告サインを出して欲しい。
もしかしたら警告を出しているのに、偶然その時、お酒を飲んだりしていて、警告を無視しているのかもしれませんね。
とまあ、言い訳ですが。
最近、PacBio社が学会やセミナーのスライドショーを立て続けに公開しているので、これは是非皆さんに紹介したいと思いました。興味のある方はご覧ください。
9月、PacBioユーザーグループミーティング(UGM)
10月、アメリカ人類遺伝学会(ASHG)
11月、Cold Spring Harbor Laboratory, Genome Informatics(CSHL)
での発表スライドです。
UGMは、一部、非公開のスライドもあります。これは仕方ない。
まずはUGM こちらは以前ブログでも紹介しましたがようやく、PacBioのブログでも、その様子が公開されました。
小さな英語文字で少し見づらいかもしれませんが、ところどころに青色でリンクがあり、そこからプレゼンの資料がダウンロードできます。
個人的にはRNA-Seqが好きです。全長cDNAシークエンスというもので、これは最近PacBioが力をいれており、間違いなく来年、大きなブレークスルーがあると思います。
バクテリアデノボアセンブリのHGAPのように。
続いてASHG
PacBioのワークショップがありました。そのスピーカーのビデオが公開されています。
ちなみに2番目のスピーカー(Dr. Eicher)は、Pacで10X ヒトゲノム読んで公開した方です。
最後にCSHL Genome Informatics
これは私は参加していないので実際聞いていないのですが、HGAPの開発者、Jason ChinによるDiploid Assemblyの発表です。
今のアセンブラーでは二倍体に対応していません。PacBioのロングリードをもってしても、現在のCelera Assemblerは二倍体を考慮したアセンブリはできないので、PacBioではそれに代わるアルゴリズムの開発をしています。
インフォマティシャンなら要チェック! 来年くらいに論文になるでしょうか。
これも間違いなく来年の大きなブレークスルーになるでしょう。
その前に本人からこのプレゼン内容を聞いておきます。
先日、熱出しました。(前回はシンガポール帰国直後)
PacBioのシークエンサーも、内部の温度を常に監視していて、ちょっと異常があると警告メッセージを出すんですが、人間の体も具合が悪くなる前に軽い警告サインを出して欲しい。
もしかしたら警告を出しているのに、偶然その時、お酒を飲んだりしていて、警告を無視しているのかもしれませんね。
とまあ、言い訳ですが。
最近、PacBio社が学会やセミナーのスライドショーを立て続けに公開しているので、これは是非皆さんに紹介したいと思いました。興味のある方はご覧ください。
9月、PacBioユーザーグループミーティング(UGM)
10月、アメリカ人類遺伝学会(ASHG)
11月、Cold Spring Harbor Laboratory, Genome Informatics(CSHL)
での発表スライドです。
UGMは、一部、非公開のスライドもあります。これは仕方ない。
まずはUGM こちらは以前ブログでも紹介しましたがようやく、PacBioのブログでも、その様子が公開されました。
小さな英語文字で少し見づらいかもしれませんが、ところどころに青色でリンクがあり、そこからプレゼンの資料がダウンロードできます。
個人的にはRNA-Seqが好きです。全長cDNAシークエンスというもので、これは最近PacBioが力をいれており、間違いなく来年、大きなブレークスルーがあると思います。
バクテリアデノボアセンブリのHGAPのように。
続いてASHG
PacBioのワークショップがありました。そのスピーカーのビデオが公開されています。
- Characterizing Structural Variation in the Human Genome using Long Read SMRT Sequencing
- Hosted by Jonas Korlach, Ph.D., Pacific Biosciences
- Resolving Complex Regions of Genomes using Long-read Sequencing Technology
- Evan Eichler, Ph.D., University of Washington
- Highlighting Unexplored Genomic Regions with SMRT Sequencing
- Ali Bashir, Ph.D., Mt. Sinai School of Medicine
- Targeting Complex Structural Motifs in Genes And Haplotypes with Long-Read SMRT Sequencing
- Swati Ranade, Ph.D., Pacific Biosciences
ちなみに2番目のスピーカー(Dr. Eicher)は、Pacで10X ヒトゲノム読んで公開した方です。
最後にCSHL Genome Informatics
これは私は参加していないので実際聞いていないのですが、HGAPの開発者、Jason ChinによるDiploid Assemblyの発表です。
今のアセンブラーでは二倍体に対応していません。PacBioのロングリードをもってしても、現在のCelera Assemblerは二倍体を考慮したアセンブリはできないので、PacBioではそれに代わるアルゴリズムの開発をしています。
インフォマティシャンなら要チェック! 来年くらいに論文になるでしょうか。
これも間違いなく来年の大きなブレークスルーになるでしょう。
その前に本人からこのプレゼン内容を聞いておきます。
2013年10月23日水曜日
Human 10x データ公開
お待たせしました!
って、待ってないかな? Humanを10xで読んだときのデータが公開されました。
Evan Eichler 博士(Howard Hughes Medical Institute, University of Washington)との共同で、このたびPacBioでは、CHM1TERTセルラインを読みました。
詳しくはPacBioのブログにのっています。
以下は、DevNet(PacBioの別サイト)から
ちなみにまだ、この試薬はアメリカの限られたサイトでのみ使用可です。
日本ではもう少しかかりますね。
PacBioのブログに行けば、サブリードのグラフも見てとれますが、結構すごい。
15kbとか25kbとか、読めてます。
10日間、66セルを使って、ヒトゲノムの10xデータ
彼らはhg19(GRCh37)にマッピングして、Deletionなどを見つけています。
ところで今、BostonのASHGに来ているのですが、ちょうどさっきGRCh38はこうなる!みたいな話を聞きました。
37よりもCentromere や、Gapのデータが更新されているそうです。
1000人ゲノムデータやOptical Mappingのデータも使ってかなりの更新になるそうです。
くわしくは彼らのブログをチェック!
このPacのデータも、GRCh38にマップしてみたい!
ところで、先ほどの10xのPacデータ、Pacブログのところに書いてある通り、ダウンロードできます。
メールを送ると自動でURLが帰ってきますので、興味ある方はどうぞ。
マッピングデータ(BAM)だけでも価値はあるかも。
って、待ってないかな? Humanを10xで読んだときのデータが公開されました。
Evan Eichler 博士(Howard Hughes Medical Institute, University of Washington)との共同で、このたびPacBioでは、CHM1TERTセルラインを読みました。
詳しくはPacBioのブログにのっています。
以下は、DevNet(PacBioの別サイト)から
Sequencing Data Statistics
Total number of reads: 3,679,463
Total number of post-filtered bases: 32,559,803,198
Read length statistics
Half of sequenced bases in reads greater than: 10,985 bp
5% of reads longer than: 19,060 bp
SMRTbell template statistics
Longest DNA insert sequenced: 41,460 bp
5% of sequenced DNA inserts longer than: 18,060 bp
Average sequenced DNA insert length: 7,406 bp
PacBio RS II instrument time for sequencing: 10 days
Number of SMRT Cells: 66ちなみにまだ、この試薬はアメリカの限られたサイトでのみ使用可です。
日本ではもう少しかかりますね。
PacBioのブログに行けば、サブリードのグラフも見てとれますが、結構すごい。
15kbとか25kbとか、読めてます。
10日間、66セルを使って、ヒトゲノムの10xデータ
彼らはhg19(GRCh37)にマッピングして、Deletionなどを見つけています。
ところで今、BostonのASHGに来ているのですが、ちょうどさっきGRCh38はこうなる!みたいな話を聞きました。
37よりもCentromere や、Gapのデータが更新されているそうです。
1000人ゲノムデータやOptical Mappingのデータも使ってかなりの更新になるそうです。
くわしくは彼らのブログをチェック!
このPacのデータも、GRCh38にマップしてみたい!
ところで、先ほどの10xのPacデータ、Pacブログのところに書いてある通り、ダウンロードできます。
メールを送ると自動でURLが帰ってきますので、興味ある方はどうぞ。
マッピングデータ(BAM)だけでも価値はあるかも。
2013年10月18日金曜日
PacBio をヒトトランスクリプトーム解析へ Nature Biotech (補足)
先日の投稿の補足です。
CCSを476,000本とるのに何セル使ったのか、どうしても気になったので著者に聞いてみました。
使用したのは35セルだそうです。
40セルくらいかな、と予想した数がまんざら外れてはいませんでした!
彼らがシークエンスした当時のマシンのバージョンは、RS I、使用試薬はXL/C2、Movieは55分 です。
今なら、RS II、P4/C2、120分Movieで読めるので、1セルあたり50,000くらいのCCSはできるだろう、というコメントをいただきました。
それなら10セル程度で、476,000本は取れるでしょう。
良い感じ。
CCSを476,000本とるのに何セル使ったのか、どうしても気になったので著者に聞いてみました。
使用したのは35セルだそうです。
40セルくらいかな、と予想した数がまんざら外れてはいませんでした!
彼らがシークエンスした当時のマシンのバージョンは、RS I、使用試薬はXL/C2、Movieは55分 です。
今なら、RS II、P4/C2、120分Movieで読めるので、1セルあたり50,000くらいのCCSはできるだろう、というコメントをいただきました。
それなら10セル程度で、476,000本は取れるでしょう。
良い感じ。
2013年10月17日木曜日
PacBio をヒトトランスクリプトーム解析へ Nature Biotech
久しぶり、3年ぶりくらいに、九州に来ています。 飛行機の窓から見える富士山はきれいでした。 羽田からだと左側の窓側に座れば、ちょうど富士山の真上を見ることができるんですよね。
冬は雪がかぶってもっときれいです。
その後、名古屋上空を飛び、関西上空を飛び、広島市上空を飛び、2時間ちょいで福岡空港です。
博多に高層ビルが無いのは、上空が飛行機の進入路になっているからなんですね。 高い建物は天神よりもっと港側に見えました。
さて、先週末、Nature Biotechに、PacBioを使用したヒトトランスクリプトームの論文が出ました。
彼らの取り組みは、「PacBioでヒトの転写産物を完全長で読むことができる」ことを示した最初の論文です。
彼らの研究は、私も部分的にはフォローしていましたが、恥ずかしながら、解析手法を誤解していました。
てっきり、ロングのサブリードを、ショートリードなどでエラー補正してからゲノムにマッピングしたと思い込んでいました。しかし彼らはCCSを使い、エラー補正しないでゲノムにマッピングしています。
エラー補正をするということは、ショートリードのマッピングバイアスがかかる、ということです。
これは厳密にはPacBioの良さ(リピートに強い、GC含量によらない、一定のカバレッジ、などの特徴)を打ち消してしまうことになります。 彼らもそれを述べていて、エラー補正のいらないほど十分に精度の高い、CCSを使うことにしたそうです。
CCSは、デフォルトでは、ライブラリサイズの2パス以上読んだときの、そのコンセンサス配列です。
現在は、5~6パス程度で99%の精度になります。
「デフォルトでは」と書いたのは、間もなくリリースされる二次解析ソフトウェアSMRT Analysis 2.1 では、ユーザが自由にパスの条件を変えることができるからです。
それはさておき、彼らはCCSを採用した。
その数476,000 相当な数です。
彼らは長さで分けてライブラリを作らずに、(サイズセレクションをせずに)、全ての種類の転写産物を一度に読んでいます。 CCSの平均長は1kbだったそうです。
CCSは2パス以上読んだときのコンセンサスですので、CCSの平均長が1kbだとすると、最低2kb以上は読めている必要があります。
彼らが実験をしたときはXL/C2試薬で、55分Movieで2回(合計15万ZMW)読んでいますので、およそ、平均3kbの長さのリードを読めていると思います。
これも経験上の想像ですが、1セルあたり15万ZMWの30%からちゃんとしたデータが出て、出力5万リード、さらにそのうち2kb以上のリードが6割くらいあったとすると12,000本リード。これがCCSとなります。
1セルあたり12,000CCSと仮定すると、476,000CCSを出すのには、40セル必要になるわけで、これはちょっと、計算を誤った感あり。
もちろん今は、P4/C2試薬といって、XL/C2よりも精度・スループットともに向上していますので、このセル数の推定は正しくはありません。
しかし、CCSを使うとなると、それだけスループットを犠牲にしなければならないのは変わりません。
そういうわけで、今後出てくる論文は、CCSよりもサブリード、それもエラー補正したサブリード、を主役に持ってくるはずです。
私は個人的には、CCSに期待しています。それは先の理由で、ショートリードで補正したらショートのバイアスがかかってしまうからです。
最後にライブラリの話を少しします。
PacBioが公開している「今の」cDNAプロトコルでは、ライブラリを3種類に分けることを推奨しています。
cDNAにしたあと増幅してゲルに流して、1.5kb未満、1.5kbから3.0kb、3.0kb以上、という3種類のサイズに分けてからライブラリを作り、それぞれ別々に読みます。 これは、ローディングバイアスという、PacBioのSMRT Cell独自の性質を回避するためです。
短いライブラリ程ZMWに入りやすいので、結果として短いライブラリばかり読まれてしまう、という現象を、ローディングバイアスといいます。
これを防ぐために、ライブラリサイズを3つに分けて、別々のセルで読むのです。
しかし今回の論文では、これを行っていません。
CCSの多くが1.5kb未満だったという結果は、ローディングバイアスによるものなのか、ほとんどの転写産物は1.5kb未満という知見に一致するからこれが真実なのか、私にはいまひとつはっきりわかりません。
ちなみに、454のデータとも比較しており、PacBioで読んだ方がGENCODEの登録遺伝子をより多くカバーしていた、という嬉しい結果も書かれていました!
2013年9月30日月曜日
PacBio RS II によるバクテリアゲノムアセンブリ:海外トップユーザの状況から
今日は久しぶりに会社のチームみんなで集まってビアガーデンパーティをしました。
出張が多いメンバーが一度にみんなそろうことはめったに無いのです。
9月30日、ビアガーデンとしてはちょっと肌寒い時期でしたが、大手町サンケイビル4階の「天空のビアガーデン」
今年の夏を締めくくるにふさわしい、とても良いひと時でした。
さてさて、バクテリアサイズのゲノムのデノボアセンブリには、PacBio RSを超える機械はこの世に存在しない! というのはこのブログを読んでいる皆様にはもう常識?ですが、海外でももちろんその通りです。
JGI(Joint Genome Institute)という、アメリカエネルギー省管轄の研究所がカリフォルニア州ウォルナットクリークにありまして、ここでは2台のPacBio RSがフル活動しています。
エネルギー省の施設なので、読んでいるバクテリアの種類は、病原菌というよりもどちらかというと環境や生態系に関する重要バクテリアや、バイオ燃料開発などで重要なバクテリアで、これらをがんがん読んでいるそうです。
彼らのホームページにもPacBioシークエンサーのことが書かれていて、HGApのことについても触れられています。HGApはもうご存知、PacBioのロングリードだけでエラー補正からアセンブリ、Contig Polishingまでを自動で行うパイプラインのことですね。
このアルゴリズムの開発にも、JGIは大きな貢献をしました。
http://www.jgi.doe.gov/News/news_13_05_06.html
さて、JGIでは、大量のサンプルをガンガン読んでいるために、サンプル調整を手で行うのは大変になりました。
そこで導入したのが、PerkinElmer社から売られている自動分注装置。
Caliper社のScicloneという自動サンプル調整ロボット機械には、PacBio用のアプリケーションが作られ、その名も Maestro PacBio 10Kb DNA Library Prep
マエストロですよ!好きです、こういう自身満々の名前
http://www.perkinelmer.com/Catalog/Family/ID/Sciclone%20NGS%20Workstation
この装置は、PacBioの3Kb - 10Kbのライブラリ調整を自動で行ってくれるそうです。
スループットは一度に8 から 96 サンプルで、8時間以内で終了するそうです。
このような装置は、いろんな種類のサンプルを読む場合、たくさんのライブラリを作る必要があるので、大活躍するかもしれませんね。
反対に、大型ゲノムを数種類だけ読むような場合、最初にライブラリを作ってしまえばあとはできているライブラリをたくさん読むだけなので、サンプル調整は手で行った方が安上りかもしれません。
いずれにせよ、たくさんの種類のバクテリアを読んでいるJGIには、この手の機械が有用なのでしょう。
これがその自動化マシンを使ったときの、DNAサンプルからContigデータまでの流れです。
PacBioのプレゼンより引用
DNAのシェアリングまではG-tubeでやるんですね。ここはマニュアルかあ。
断片化した後の、ライブラリ調整を、PacBioプロトコルに合わせてこの装置がやってくれるわけですね。
出来上がったライブラリをシークエンサーに乗せるわけですが、サイズセレクションは当然これもマニュアルでしょうね。
回収率はどれくらいなんでしょう?
出張が多いメンバーが一度にみんなそろうことはめったに無いのです。
9月30日、ビアガーデンとしてはちょっと肌寒い時期でしたが、大手町サンケイビル4階の「天空のビアガーデン」
今年の夏を締めくくるにふさわしい、とても良いひと時でした。
さてさて、バクテリアサイズのゲノムのデノボアセンブリには、PacBio RSを超える機械はこの世に存在しない! というのはこのブログを読んでいる皆様にはもう常識?ですが、海外でももちろんその通りです。
JGI(Joint Genome Institute)という、アメリカエネルギー省管轄の研究所がカリフォルニア州ウォルナットクリークにありまして、ここでは2台のPacBio RSがフル活動しています。
エネルギー省の施設なので、読んでいるバクテリアの種類は、病原菌というよりもどちらかというと環境や生態系に関する重要バクテリアや、バイオ燃料開発などで重要なバクテリアで、これらをがんがん読んでいるそうです。
彼らのホームページにもPacBioシークエンサーのことが書かれていて、HGApのことについても触れられています。HGApはもうご存知、PacBioのロングリードだけでエラー補正からアセンブリ、Contig Polishingまでを自動で行うパイプラインのことですね。
このアルゴリズムの開発にも、JGIは大きな貢献をしました。
http://www.jgi.doe.gov/News/news_13_05_06.html
さて、JGIでは、大量のサンプルをガンガン読んでいるために、サンプル調整を手で行うのは大変になりました。
そこで導入したのが、PerkinElmer社から売られている自動分注装置。
Caliper社のScicloneという自動サンプル調整ロボット機械には、PacBio用のアプリケーションが作られ、その名も Maestro PacBio 10Kb DNA Library Prep
マエストロですよ!好きです、こういう自身満々の名前
http://www.perkinelmer.com/Catalog/Family/ID/Sciclone%20NGS%20Workstation
この装置は、PacBioの3Kb - 10Kbのライブラリ調整を自動で行ってくれるそうです。
スループットは一度に8 から 96 サンプルで、8時間以内で終了するそうです。
このような装置は、いろんな種類のサンプルを読む場合、たくさんのライブラリを作る必要があるので、大活躍するかもしれませんね。
反対に、大型ゲノムを数種類だけ読むような場合、最初にライブラリを作ってしまえばあとはできているライブラリをたくさん読むだけなので、サンプル調整は手で行った方が安上りかもしれません。
いずれにせよ、たくさんの種類のバクテリアを読んでいるJGIには、この手の機械が有用なのでしょう。
これがその自動化マシンを使ったときの、DNAサンプルからContigデータまでの流れです。
PacBioのプレゼンより引用
DNAのシェアリングまではG-tubeでやるんですね。ここはマニュアルかあ。
断片化した後の、ライブラリ調整を、PacBioプロトコルに合わせてこの装置がやってくれるわけですね。
出来上がったライブラリをシークエンサーに乗せるわけですが、サイズセレクションは当然これもマニュアルでしょうね。
回収率はどれくらいなんでしょう?
ラベル:
PacBioを使った研究の論文,
技術,
実験,
装置
2013年9月29日日曜日
公式アナウンスメント: Roche Diagnostics と協力して診断機械開発へ
http://investor.pacificbiosciences.com/releasedetail.cfm?ReleaseID=793199
この発表はご覧になった方もいるかと思います。
Roche Diagnosticsと言えばみなさんご存じ、454 GS FLX、GS Junor のメーカーですね。
スイスに本社を置くRoche社は、2012年現在全世界で82,000人の社員を抱え、R&Dに80億スイスフラン(約8672億円)を投資し、455億スイスフラン(約4兆9322億円)を売り上げる、世界最大のバイオテックカンパニーです。
診断の分野でも大きなシェアを持っているそうです。
そんなRocheとPacific Biosciencesが協力して、ヒトIn vitro診断機器を開発する、というアグリーメントを結びました。
おそらく、噂が立つと思いますので先に、これだけはお伝えしておきます。
RocheがPacBioを買収するという話ではありません!!
アグリーメントの内容は、このようなものです。
- PacBioは、ヒト in vitro 診断のための新しい機械をRocheのために開発する
- 新しい機械は、PacBioのSMRTテクノロジーを使用する
- Rocheはこの新しい機械を全世界で独占販売する権利を有する
- 開発資金として、3500万ドルをRocheから受け取り、さらに4000万ドルを受けとる予定である
- PacBioは、ヒト in vitro 診断以外の分野では、これまでどおり機械の開発・販売を続ける
さっそくいろんな噂がツィートされているみたいです。
でも、噂の類はここには書きませんので、ご安心ください。
2013年9月25日水曜日
アメリカユーザーグループミーティングin Palo Alto
たまにチェックするブログがあります。 (http://www.homolog.us/blogs/blog/2013/09/21/end-illumina-era)
ここに、先週、カリフォルニア州パロアルトで行われた「北米・PacBioユーザーグループミーティング」の内容が書かれています!
私も今回は参加したのでわかりますが、このブログはすごく良くまとまっているので一見の価値ありです!
「ショートリードの時代の終わり?」と題していますが、バクテリアデノボの世界ではまさにそうでしょう。
今回のグループミーティングでも、バクテリア関連(デノボアセンブリ、メチレーション)が4割、トランスクリプトームが3割、高等生物アセンブリが2割、MHCなどその他が1割、という割合だった気がします。
およそ60人が参加した今回のグループミーティング(休憩時間とランチタイムの様子)
さて、私が興味を持ったトピックスは、バクテリア以外です。
1.Arabidopsis(Salk Institute for Biological Studies);Arabidopsisは、Colombia 10のゲノム配列が読まれ公開されていますが、まだ800株ほどの読まれていない株があるそうです。
彼らはPacBioのリードだけを用いてアラビの株をたくさん読んで、株間のゲノム比較を行っています。
Ler-0 株を例にとると、20kbライブラリを作り、ゲノムサイズ120Mbの50x分のデータを得ました。
HGApを使ってアセンブリしたところ、Contig本数は540本、塩基総数は124Mbp、N50 = 6.2Mbp、Max 13Mbpという結果を得ました。
この、20kbライブラリ作製のところで、BluePippinを使っています。
Col0株も読んでおり、TAIR10のリファレンスゲノムとの比較では99%以上の一致を得たので、Ler-0株もそれなりに高い精度のContigが作られているという感触を持っているみたいです。
現在多くのSNPやInDelが見つかっているのでそれらを検証しているとのこと。
1株のアセンブルに要した時間はおよそ10日。うち9日間はHGApのPre-Assembly(最初のエラーコレクション)に要した時間です。 Pre-Assemblyの時間を短くするのが課題ですね。
実はSMRT Analysisの次のバージョンで、Pre-Assemblyが劇的に速くなるオプションがつくのですが、まだベータ版です。
2.チキンRNA-Seq(Gladstone Institute);ニワトリの心臓とヒトの心臓は構造が似ているため、モデルにはちょうど良いそうです。 しかしニワトリのRefSeq情報はまだ6,500配列しかなく、不十分。 そこで完全長cDNA=Isoformの配列データを得る必要があるという。
今までショートリードで読み、Trinityなどのツールを使って解析してきたが、今回PacBioを使ってIsoformデータを得ることに挑戦したそうです。
0-1kb,1-2kb,2-3kb,>3kbの4種類のライブラリを作り、フルパスサブリード配列のみを対象に、イルミナのリードでエラーコレクションし、ゲノム参照配列にアラインしました。 Geneアノテーションを行ったところ、新しいTranscriptが2000程見つかったそうです。
3.ヒトTranscriptome(Stanford Univ);LSCという配列圧縮/エラー補正アルゴリズムを用いて、完全長cDNAの解析を行っていました。スプライシングの検出には完全長が不可欠です。 曰く、Cufflinksなどのショートリード向けのツールは、ベースにしている理論が数学的でありバイオロジカルな現象を反映して無い、と。
エラーコレクションを行った後のPacBioリードを、BLATでゲノムにアラインしました。 8,084本の既知の転写産物+5,459本の新規転写産物を得たそうです。
PacBioでは間もなく、新しい試薬がリリースされる予定です。
P5/C3 ケミストリーは、ロングリードをさらに伸ばして、平均8,500bpを実現します。
と言っても、これはポリメラーゼリード長です。 サブリード長を伸ばすには、20kbライブラリ+サイズセレクションが必須です。
そんなわけで、今回の演者で20kbライブラリを作った人はみな、BluePippinを使ったサイズセレクションを行っていました。
BPのメーカーであるSage Science社のTwitterで知ったのですが、MRSAから抽出したDNAを、20kbライブラリ+BPサイズセレクション実行後、P5/C3試薬を使い3時間Movieで読んだら、最高34,500bpのポリメラーゼリードを得たそうです!
ここに、先週、カリフォルニア州パロアルトで行われた「北米・PacBioユーザーグループミーティング」の内容が書かれています!
私も今回は参加したのでわかりますが、このブログはすごく良くまとまっているので一見の価値ありです!
「ショートリードの時代の終わり?」と題していますが、バクテリアデノボの世界ではまさにそうでしょう。
今回のグループミーティングでも、バクテリア関連(デノボアセンブリ、メチレーション)が4割、トランスクリプトームが3割、高等生物アセンブリが2割、MHCなどその他が1割、という割合だった気がします。
およそ60人が参加した今回のグループミーティング(休憩時間とランチタイムの様子)
さて、私が興味を持ったトピックスは、バクテリア以外です。
1.Arabidopsis(Salk Institute for Biological Studies);Arabidopsisは、Colombia 10のゲノム配列が読まれ公開されていますが、まだ800株ほどの読まれていない株があるそうです。
彼らはPacBioのリードだけを用いてアラビの株をたくさん読んで、株間のゲノム比較を行っています。
Ler-0 株を例にとると、20kbライブラリを作り、ゲノムサイズ120Mbの50x分のデータを得ました。
HGApを使ってアセンブリしたところ、Contig本数は540本、塩基総数は124Mbp、N50 = 6.2Mbp、Max 13Mbpという結果を得ました。
この、20kbライブラリ作製のところで、BluePippinを使っています。
Col0株も読んでおり、TAIR10のリファレンスゲノムとの比較では99%以上の一致を得たので、Ler-0株もそれなりに高い精度のContigが作られているという感触を持っているみたいです。
現在多くのSNPやInDelが見つかっているのでそれらを検証しているとのこと。
1株のアセンブルに要した時間はおよそ10日。うち9日間はHGApのPre-Assembly(最初のエラーコレクション)に要した時間です。 Pre-Assemblyの時間を短くするのが課題ですね。
実はSMRT Analysisの次のバージョンで、Pre-Assemblyが劇的に速くなるオプションがつくのですが、まだベータ版です。
2.チキンRNA-Seq(Gladstone Institute);ニワトリの心臓とヒトの心臓は構造が似ているため、モデルにはちょうど良いそうです。 しかしニワトリのRefSeq情報はまだ6,500配列しかなく、不十分。 そこで完全長cDNA=Isoformの配列データを得る必要があるという。
今までショートリードで読み、Trinityなどのツールを使って解析してきたが、今回PacBioを使ってIsoformデータを得ることに挑戦したそうです。
0-1kb,1-2kb,2-3kb,>3kbの4種類のライブラリを作り、フルパスサブリード配列のみを対象に、イルミナのリードでエラーコレクションし、ゲノム参照配列にアラインしました。 Geneアノテーションを行ったところ、新しいTranscriptが2000程見つかったそうです。
3.ヒトTranscriptome(Stanford Univ);LSCという配列圧縮/エラー補正アルゴリズムを用いて、完全長cDNAの解析を行っていました。スプライシングの検出には完全長が不可欠です。 曰く、Cufflinksなどのショートリード向けのツールは、ベースにしている理論が数学的でありバイオロジカルな現象を反映して無い、と。
エラーコレクションを行った後のPacBioリードを、BLATでゲノムにアラインしました。 8,084本の既知の転写産物+5,459本の新規転写産物を得たそうです。
PacBioでは間もなく、新しい試薬がリリースされる予定です。
P5/C3 ケミストリーは、ロングリードをさらに伸ばして、平均8,500bpを実現します。
と言っても、これはポリメラーゼリード長です。 サブリード長を伸ばすには、20kbライブラリ+サイズセレクションが必須です。
そんなわけで、今回の演者で20kbライブラリを作った人はみな、BluePippinを使ったサイズセレクションを行っていました。
BPのメーカーであるSage Science社のTwitterで知ったのですが、MRSAから抽出したDNAを、20kbライブラリ+BPサイズセレクション実行後、P5/C3試薬を使い3時間Movieで読んだら、最高34,500bpのポリメラーゼリードを得たそうです!
2013年9月24日火曜日
NGS現場の会で話したPacBio最近のアプリケーション
知らなかったのですが、こんなのがあるそうです。
日本にある、論文にはなっていないけれどお助けツールとして広く知られている、役に立っている、(基本無料で公開している)ライフサイエンス関連の情報についてのグランプリ賞です。
データベース部門、ソフトウェア部門、ウェブ部門、とあって、恥ずかしながら自分でも知らないものもありました。
そこに私のブログが2つ、ノミネートしているのを発見! おおっ!
投票締め切りは9月27日、急げ!
それはさておき、「NGS現場の会」でお話しした、最近のPacBioのアプリケーションを紹介します。
まずは「パンドラウイルス」
今年の7月に公開された、ゲノムサイズがウイルスのくせに2.5MBもある巨大ウイルスの、ゲノム解読に、PacBioが使われました。
論文になっています。 解析の詳細は、サプリメントに書いてありますので、一読をお勧めします。
もっとも、PacBioだけではなく、Illuminaと454でも読んでいて、それぞれでContigを作ったあと、いったんPhrapでマージし、その後、PacBioのロングリードでScaffoldを作ったり、Gapを埋めたり、しています。
面白いのが最後に、ContigをIlluminaデータでエラーコレクションしていること。
エラーコレクションと言えば、アセンブリする前にすることしか私の頭になかったのですが、ScaffoldingにPacBioリードを使うのであれば、後でコレクションするのもアリなんですよね。
続いて、「100K Foodborne Pathogen Genome Project 」
去年から行われているこのゲノムプロジェクトは、食中毒原因菌を100,000種類(株)読んで、ゲノム配列やメチレーションのデータを公開しています。
サルモネラ菌、リステリア菌、大腸菌、など5種あまりを優先的に読んでいて、今後はもっと広げていく予定です。
このプロジェクトでは、PacBioだけで読んだりしているゲノムがあります。
2013年7月に、20種類のゲノム解読を完了して公開しました。
最近の情報では今年の秋に、1,500種類を完了して公開するそうです。
ちなみに、このプロジェクトには、PacBioのほかにもいくつかの企業が参加していて、CLC-BIOの名前もありました。
続いて「ハイブリッドHGApによる大型ゲノムアセンブリへの応用」
ゲノムサイズが350MBあるアブラムシゲノムアセンブリに、PacBioのリードを使ってHGApしたのですが、ユニークなのが、最初のPre-Assemblyに、PacBioロングリードだけではなく、IlluminaでアセンブリしたContigをも用いていることなんです。
最新バージョンのHGApでは、PacBioロングリードだけではなくて、任意のFASTQファイルも、Pre-Assembly(エラーコレクションステップ)に使えるのです。
イリノイ州立大学のチームによるこの結果は、間もなく論文になるでしょう。
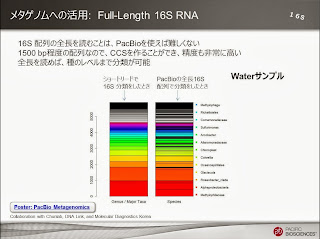
「完全長の16S配列」も、PacBioロングリードならではのアドバンテージが生かせる分野です。
1500bpくらいの配列なので、CCSでも十分よめる長さです。
韓国のチームによって出された結果は、今年のアメリカでの微生物学会にてポスター発表が行われました。そのポスターは再配布可能(というか「宣伝して」と言われている)なので、もし欲しい方がいらっしゃればお知らせください。
日本にある、論文にはなっていないけれどお助けツールとして広く知られている、役に立っている、(基本無料で公開している)ライフサイエンス関連の情報についてのグランプリ賞です。
データベース部門、ソフトウェア部門、ウェブ部門、とあって、恥ずかしながら自分でも知らないものもありました。
そこに私のブログが2つ、ノミネートしているのを発見! おおっ!
投票締め切りは9月27日、急げ!
それはさておき、「NGS現場の会」でお話しした、最近のPacBioのアプリケーションを紹介します。
まずは「パンドラウイルス」
今年の7月に公開された、ゲノムサイズがウイルスのくせに2.5MBもある巨大ウイルスの、ゲノム解読に、PacBioが使われました。
論文になっています。 解析の詳細は、サプリメントに書いてありますので、一読をお勧めします。
もっとも、PacBioだけではなく、Illuminaと454でも読んでいて、それぞれでContigを作ったあと、いったんPhrapでマージし、その後、PacBioのロングリードでScaffoldを作ったり、Gapを埋めたり、しています。
面白いのが最後に、ContigをIlluminaデータでエラーコレクションしていること。
エラーコレクションと言えば、アセンブリする前にすることしか私の頭になかったのですが、ScaffoldingにPacBioリードを使うのであれば、後でコレクションするのもアリなんですよね。
続いて、「100K Foodborne Pathogen Genome Project 」
去年から行われているこのゲノムプロジェクトは、食中毒原因菌を100,000種類(株)読んで、ゲノム配列やメチレーションのデータを公開しています。
サルモネラ菌、リステリア菌、大腸菌、など5種あまりを優先的に読んでいて、今後はもっと広げていく予定です。
このプロジェクトでは、PacBioだけで読んだりしているゲノムがあります。
2013年7月に、20種類のゲノム解読を完了して公開しました。
最近の情報では今年の秋に、1,500種類を完了して公開するそうです。
ちなみに、このプロジェクトには、PacBioのほかにもいくつかの企業が参加していて、CLC-BIOの名前もありました。
続いて「ハイブリッドHGApによる大型ゲノムアセンブリへの応用」
ゲノムサイズが350MBあるアブラムシゲノムアセンブリに、PacBioのリードを使ってHGApしたのですが、ユニークなのが、最初のPre-Assemblyに、PacBioロングリードだけではなく、IlluminaでアセンブリしたContigをも用いていることなんです。
最新バージョンのHGApでは、PacBioロングリードだけではなくて、任意のFASTQファイルも、Pre-Assembly(エラーコレクションステップ)に使えるのです。
イリノイ州立大学のチームによるこの結果は、間もなく論文になるでしょう。
「完全長の16S配列」も、PacBioロングリードならではのアドバンテージが生かせる分野です。
1500bpくらいの配列なので、CCSでも十分よめる長さです。
韓国のチームによって出された結果は、今年のアメリカでの微生物学会にてポスター発表が行われました。そのポスターは再配布可能(というか「宣伝して」と言われている)なので、もし欲しい方がいらっしゃればお知らせください。
2013年9月15日日曜日
Pacすごいぜ論文と、最新20kbサンプルデータ
Koren et.al. Reducing assembly complexity of microbial genomes with single-molecule sequencing. Genome Biology 2013, 14:R101
(http://genomebiology.com/2013/14/9/R101/abstract)
この論文は9月13日にPublishされました。
日々、PacBioのプレゼン等でこういう「Pacすごいぜ」的な情報に慣れきっている私には、それほど驚くほどのものではない、と言っては著者に失礼ですが、PacBioを知らない方やこれから学ぼうとしている方には良い参考になると思います。
ちなみに、著者は、エラー補正アルゴリズム(pacBioToCA)の作者でもあります。
さて、先々週の「NGS現場の会」の続きです。
「DeNovoの達人」で東大・笠原さんの解析フローが、公開されました。
こちら(https://cell-innovation.nig.ac.jp/wiki/tiki-index.php?page=Sprai)
デモデータで使用できるPacBioデータのリンクもありますので是非どうぞ。
E.coli 20kbのライブラリのデータを使うと、10kb以上の長さのContigは1つだけになったそうです。
詳しい手順は上記のサイトをご覧ください。
私がHGAP-CA-Quiverのフローで行ったところ、同じく1本になりました。
最初のPre-Assemblyステップで8,103bpをSeedリードに、それ以下の短いリードを補正用に使います。
そうして作られた12,418本の補正後ロングリード、総塩基数109MbpをCelera Assembly
その後QuiverでContigをPolishして得た最終Contigが、長さ4,665,426bp
その過程で作られる、サブリードの長さの分布がこちら
うーん、これはチャンピオンデータだな。
ここまで良いデータはなかなか出ないです。
10kp以上のサブリードだけでも100Mb(総塩基数)= 20X ある!
実際、20kbのライブラリをうまく作るのがキーで、サイズセレクションは必須、ということはたくさんDNAが取れることが必須、なわけですね。
2013年9月8日日曜日
「NGS現場の会」が終わって 反省(マイクの音量・声が小さかった?)
みなさんも参加されましたでしょうか?
9月4日、5日の「NGS現場の会・第三回研究会」 in 神戸
私は3日の夜に入り、いつものことながらプレゼンの練習と最終確認を深夜2時までやっていました。
ちなみに泊まったホテルは「ヴィラフォンテーヌ」という、名前は発音しずらいけれど6000円前後ですごく良い部屋&朝食が付いてくる!(http://www.hvf.jp/sannomiya/) 三ノ宮ではここおすすめ
さて、4日はいろんな発表聞いて、たくさんの方と話して、懇親会後も大勢で飲みに行って、と、現場の人間の集まりにふさわしい一日でした。
私の発表は次の日。 もちろん早起きできるはずもなく、しかし9時55分からの発表なので9時20分ごろには会場入りしました。
あとで知ったのですが、8時からのモーニングセッション「いまさら聞けない NGS超!入門」(株式会社ジナリス竹田 綾 さん)にて、このブログを紹介してくださったそうです。 ありがとう!!
私の発表は、ギャグも受けて良かった良かった。 ロードマップの話はみなさん驚いていたようで。
そしてフリーシークエンスキャンペーンの発表! <--こちら詳しくは現場の会のメーリングリストに流した後、本ブログでも紹介する予定です。
ちなみに私の話の内容は、こんな感じ
----------------------------------------------------------------------
聞き逃した方もいらっしゃると思いますので、内容についてはこのブログでも書こうと思います。
まず最初に、現在のスループットで、これだけ良い結果が出るという例を紹介します。
これは私たちが6月に、大腸菌のゲノムで20kbのライブラリを作り、それをBlue Pippin(TM)でサイズセレクションし、1セルで読んだ時のデータです。
100bp以上でフィルタリングすると平均リード長が6,149 bp
参照配列(K12-MG1655)にマッピングした時のサブリード平均長が4,968 bp
できたコンセンサスと、リファレンスとの一致は99.9967%
その時のカバレッジは86.99
このデータで示される通り、精度は高い! 99.9967%はすごいと思います。
さらに、このリードだけ(1 SMRT Cell だけのデータ)でアセンブルして、見事1本のContigができ、これの参照配列とのドットプロットはご覧の通りです。 ほとんど一緒!
私の発表の後、同じ会場で、「DeNovoの達人」という企画がありました。
これはもう、すごい!の一言。
阪大微研さんで、MiSeq、Ion Torrent、GS Jr、PacBioを使用して、同じゲノムを読んでもらい、そのデータを使って、3名の達人たちにアセンブリ解析してもらった結果を公開するという企画です。
皆さん、得意な分野で工夫して解析していました。
この企画、ツイートOKだったかどうか忘れたので、詳細は書きませんが、結論だけ言うとPacBioの大勝利でした!
もちろん、解析して頂いた東大・笠原さん(Master of DeNovo)のおかげです。
ゲノムサイズも数も知らされず、ドライだけで見事、サイズと数を、答えと一致させたのですから。
ロングリードでないと絶対つながらない配列は、ショートリードでいくら読んでもつながらないのです。
この会場、私のPacBio発表の後に、最終的にPacBioの宣伝になった「DeNovoの達人」、があったわけです。
これは偶然です。 本当に偶然です。
私は、「DeNovoの達人」の結果は、前もって知らされていませんでした。 だから結果についてはちょっと心配していたくらいです。
と、2日間、非常に充実していました。 スタッフのみなさん、お疲れ様でした。
神戸はいいところですね。
9月4日、5日の「NGS現場の会・第三回研究会」 in 神戸
私は3日の夜に入り、いつものことながらプレゼンの練習と最終確認を深夜2時までやっていました。
ちなみに泊まったホテルは「ヴィラフォンテーヌ」という、名前は発音しずらいけれど6000円前後ですごく良い部屋&朝食が付いてくる!(http://www.hvf.jp/sannomiya/) 三ノ宮ではここおすすめ
さて、4日はいろんな発表聞いて、たくさんの方と話して、懇親会後も大勢で飲みに行って、と、現場の人間の集まりにふさわしい一日でした。
私の発表は次の日。 もちろん早起きできるはずもなく、しかし9時55分からの発表なので9時20分ごろには会場入りしました。
あとで知ったのですが、8時からのモーニングセッション「いまさら聞けない NGS超!入門」(株式会社ジナリス竹田 綾 さん)にて、このブログを紹介してくださったそうです。 ありがとう!!
私の発表は、ギャグも受けて良かった良かった。 ロードマップの話はみなさん驚いていたようで。
そしてフリーシークエンスキャンペーンの発表! <--こちら詳しくは現場の会のメーリングリストに流した後、本ブログでも紹介する予定です。
ちなみに私の話の内容は、こんな感じ
----------------------------------------------------------------------
PacBioシークエンサーの基礎・特徴
PacBio Q&A
メチレーション検出の現状
サンプルプレップの全体像
塩基の損傷がシークエンスに与える影響
メチレーション検出の現状
サンプルプレップの全体像
塩基の損傷がシークエンスに与える影響
最近の注目トピックス
パンドラウイルスのゲノム解読
100K ゲノムプロジェクト
大型ゲノムアセンブリ
全長16Sシークエンス
全長HLAシークエンス
パンドラウイルスのゲノム解読
100K ゲノムプロジェクト
大型ゲノムアセンブリ
全長16Sシークエンス
全長HLAシークエンス
最後に
ロードマップとキャンペーンの紹介
-----------------------------------------------------------------------聞き逃した方もいらっしゃると思いますので、内容についてはこのブログでも書こうと思います。
まず最初に、現在のスループットで、これだけ良い結果が出るという例を紹介します。
これは私たちが6月に、大腸菌のゲノムで20kbのライブラリを作り、それをBlue Pippin(TM)でサイズセレクションし、1セルで読んだ時のデータです。
100bp以上でフィルタリングすると平均リード長が6,149 bp
参照配列(K12-MG1655)にマッピングした時のサブリード平均長が4,968 bp
できたコンセンサスと、リファレンスとの一致は99.9967%
その時のカバレッジは86.99
このデータで示される通り、精度は高い! 99.9967%はすごいと思います。
さらに、このリードだけ(1 SMRT Cell だけのデータ)でアセンブルして、見事1本のContigができ、これの参照配列とのドットプロットはご覧の通りです。 ほとんど一緒!
私の発表の後、同じ会場で、「DeNovoの達人」という企画がありました。
これはもう、すごい!の一言。
阪大微研さんで、MiSeq、Ion Torrent、GS Jr、PacBioを使用して、同じゲノムを読んでもらい、そのデータを使って、3名の達人たちにアセンブリ解析してもらった結果を公開するという企画です。
皆さん、得意な分野で工夫して解析していました。
この企画、ツイートOKだったかどうか忘れたので、詳細は書きませんが、結論だけ言うとPacBioの大勝利でした!
もちろん、解析して頂いた東大・笠原さん(Master of DeNovo)のおかげです。
ゲノムサイズも数も知らされず、ドライだけで見事、サイズと数を、答えと一致させたのですから。
ロングリードでないと絶対つながらない配列は、ショートリードでいくら読んでもつながらないのです。
この会場、私のPacBio発表の後に、最終的にPacBioの宣伝になった「DeNovoの達人」、があったわけです。
これは偶然です。 本当に偶然です。
私は、「DeNovoの達人」の結果は、前もって知らされていませんでした。 だから結果についてはちょっと心配していたくらいです。
と、2日間、非常に充実していました。 スタッフのみなさん、お疲れ様でした。
神戸はいいところですね。
2013年7月27日土曜日
共生菌ゲノムの論文、日韓ほぼ同時Published
先週の木曜日、品川の京都大学東京オフィスにて、「NGS現場の会・第三回研究会」のスポンサー企業ミーティングがあり、行ってきました。
現場の会の集まりだから、わざと、アロハシャツ着て行ったら案の定、司会の渡辺さんにいじられましたよ。
久しぶりに会ったひとも何人かいて、懇親会の後も飲みに行ったり。
みんなで現場の会を盛り上げていきましょう!
さて、今月、アジアから2本、PacBioユーザの論文がPublishされました!
1つは日本、1つは韓国です。
- 日本:Shibata TF et al., Complete Genome Sequence of Burkholderia sp. Strain RPE64, Bacterial Symbiont of the Bean Bug Riptortus pedestris. Genome Announc. 1(4):e00441-13.
- 韓国:Seung Chul Shin et al., Advantages of Single-Molecule Real-Time Sequencing in High-GC Content Genome. PLoS ONE 8(7): e68824
HiSeq2000で何種類かのペアエンドライブラリを読み、PacBioで6kbライブラリを読み、Allpaths-LGでハイブリッドアセンブリしています。
こちらは南極圏に住む地衣類の一種、Cladonia borealisの共生菌、Streptomyces sp. PAMC 26508のゲノムを読みました。
PacBioは8kbライブラリをロングリード用に、1.5kbライブラリをCCS用に読み、そのほか300bpペアエンドをIllumina HiSeq2000で、7kbペアエンドをGS-FLXで、読んでいます。
pacBioToCAを使ったエラーコレクションの後で、Celera Assemblyしています。
エラー補正に使うリードの種類で、どれだけアセンブリの成果が変わるか、見るとへえー、って感じです。
ところで、「NGS現場の会・第三回研究会」で、ぜひ、注目してほしい企画があります!
その名も、「DeNovoの達人」
同じサンプルを、PacBio、GS Junior、MiSeq、PGMで阪大微研さんで読んで頂きました。 これを、東大、慶応大、OISTのエキスパートがアセンブルに挑戦する!という企画。
このポスター好きです。作ったひとセンス良い!
2013年7月18日木曜日
PacBioの使い方・質問 募集!
ANAのボーイング747型機(通称ジャンボ機)は、来年3月で退役するそうです。ANAでは現在4機所有していて、どれも20年くらい働いているとのこと。 機長のアナウンスで知りました。
今回初めて、2階席に乗ったのですが、意外と広くて、窓側席には小物がしまえるスペースもあり、圧迫感が無いのにびっくりです。 でも、あと何回乗れるかなあ。
東海道山陽新幹線の500系車両、ってわかりますか? 青いカラーのとんがりノーズで、最初に時速300㎞の営業運転を始めた新幹線です。 壁と天井が300系や700系、N700系とは違って、半円形に近いんです。 ちょうどジャンボ機の2階席のように。
この500系も好きだったんですが、何年前でしょうか、のぞみ号から引退して「こだま」になってから乗らなくなりました。
出張の楽しみと言えば、こういう移動手段を選ぶときです。 あとは、ホテルを選ぶときと食べもの屋&飲み屋を選ぶとき。 もちろん仕事第一ですよ。
さて、NGS現場の会が9月の4日と5日にあります。 そこの企業セッションをチェック!
今年もPacBioのネタを喋りますが、単純なプレゼンではつまらん、と某広報委員長さまから言われましたので、少し志向を変えて、会場の方からの意見や質問も混ぜた感じのインターラクティブなものにしたいと考えています。
そこですでに現場の会メーリングリストを通じて、「PacBioをこんなふうに使えないのか?」とか「私はこんなアイデアがある!」とか「PacBioに関連してこんな噂を聞いたのだが本当か?」といった声を募集しました。
まだ受け付けていますので、是非、こちらまでメールでお送りください。
Addressは、ken_osaki「アット」digital-biology.co.jp
ちゃんとPacBio本社に確認してお答えします。
とは言っても質問自体も思いつかないよ、という方のために、今のPacBioシークエンサーのおさらいを少し。
スループットのグラフは以下の通りです。 横軸がリード長、縦軸がリード本数です。
【リード長は一定ではない】
平均4,600bpというのは、このような分布のリード長すべての平均です。 最長リードは20,000bpを超えますが、全体からするとごく一部です。
また、リードの長さとサブリードの長さは一致しません。
【ライブラリの長さも一定ではない】
10kbのライブラリを作りたくて、ゲノムDNAをシェアリングしたとしても、6kbの長さや3kbの長さのDNAも、最終的なライブラリに含まれるかもしれません。 これはライブラリ調整の過程で、AMPureのようなビーズや、BluePippinのような電圧を利用した、サイズセレクションをかけることである程度改善できますが、それでも完全に取り除くことはできないでしょう。
結果、10kbのライブラリサンプルの中には、6kbや3kbのサイズのライブラリも混在することになります。 ここで、「読まれやすさバイアス」の問題が出てきます。
【短いライブラリほど読まれやすい】
ZMWの穴には、普通、短いライブラリ程入りやすい、という物理的な性質があることが、わかっています。
Magnetic Beadsという磁気ビーズを利用して、長いライブラリを優先的に入りやすくする方法が現在デフォルトであります。
しかし、これでも実際やってみると、3kb位のライブラリならZMWの穴に入ってしまうんですね。
数百bp単位の短いライブラリを取り除くのには効果的なんでしょうが。
というわけで、10kbをターゲットとして作ったライブラリでも、3kbのライブラリがZMWの穴に入っていることは十分考えられることです。
ただ、その割合は読んでからでないと正確にはわかりません。
で、ここからがややこしいんですが、どの長さのライブラリから、どれくらいのリードが読まれたのか、この辺を知りたいと思いませんか? もしかしたら20kbのリードのほとんどは、3kbライブラリを何度も読んでいるだけかもしれない。 10kbライブラリは実は4kbくらいしか読まれていないかもしれない、などです。
この辺は普段誰も説明しないし、PacBio本社もあまり深く調べていないようなのですが、私は気になります。
それはさておき、PacBioの新しい使い方のアイデア、噂話に対する質問、等ありましたらどんどんメールでお知らせください。
2013年6月21日金曜日
150KとP4
突然ですが、今年の流行語大賞は何でしょうね?
「アベノミクス」、「今でしょう?」、この2つのどっちかじゃないか、と今から予想しています。
http://singo.jiyu.co.jp/index.htmlを見ると、ああ、そういう言葉もはやったね、と思いますが、流行語ってほとんど定着しないんですね。
アベノミクスが来年忘れられるようでは困りますが、「今でしょう?」はまさに今しか受けない、というか、そろそろ使用期限が切れるフレーズかな?
小学生の間ではまだすごいブームらしいです。
さてさて、ユーザーの方にはもちろんアナウンス済みですが、PacBioはハード・ソフトの両面でバージョンアップします(しました)。
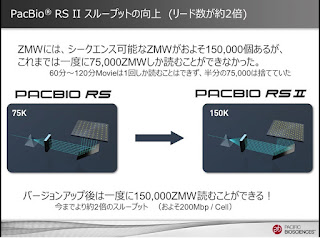
ハードは、以前「第一回アジアユーザーミーティング in シンガポール」でも触れた、RSIIです。
これは今まで、一度に読めるZMWの数が75,000だったのが、レーザーの光を分散させる光学機器の改善で、一度に150,000読めるようになったことです。
120分Movieで今まで750,000しか読めなかったのが2倍読めるようになった。つまりデータスループットが2倍になったことを意味します。
「アベノミクス」、「今でしょう?」、この2つのどっちかじゃないか、と今から予想しています。
http://singo.jiyu.co.jp/index.htmlを見ると、ああ、そういう言葉もはやったね、と思いますが、流行語ってほとんど定着しないんですね。
アベノミクスが来年忘れられるようでは困りますが、「今でしょう?」はまさに今しか受けない、というか、そろそろ使用期限が切れるフレーズかな?
小学生の間ではまだすごいブームらしいです。
さてさて、ユーザーの方にはもちろんアナウンス済みですが、PacBioはハード・ソフトの両面でバージョンアップします(しました)。
ハードは、以前「第一回アジアユーザーミーティング in シンガポール」でも触れた、RSIIです。
これは今まで、一度に読めるZMWの数が75,000だったのが、レーザーの光を分散させる光学機器の改善で、一度に150,000読めるようになったことです。
120分Movieで今まで750,000しか読めなかったのが2倍読めるようになった。つまりデータスループットが2倍になったことを意味します。
1セルあたりおよそ200Mbp、というのは控えめかもしれません。
私たちがあるバクテリアを10kbライブラリで読んでみた時、Productivity=1(信頼のあるリードデータ)が31%くらいのデータで、220Mbpでした。
その時はN=3でしたが、以来サンプルを変えて何セル読んでも大体今までの2倍は読める。
宣伝に偽り無し、でほっと一安心! (← ユーザーの立場での感想)
上図の、左側が今までのRS、右がRSII
リード長がX軸で、Y軸にその本数を示しています。 縮尺を合わせました。 どちらもProductivity=1が31%で、RSが102Mbp、RSIIが221Mbpのスループットでした。
分布「形」にはあまり差は無いのがわかるでしょう? 高さは大きく変わっています。
バージョンアップのソフトの方、こちらは、 酵素です。
P4という酵素がリリースされました。
こちらは、すごい改善! というわけでは無いのですが、より安定して、今までと同じく長く読める酵素です。
今まで、C2とXLという2種類の酵素がありました。
C2は精度は良いがスピードが遅いので長さがいまいち(平均3300bpくらい)
XLは精度が若干落ちるがスピードが速いので平均4000bpくらい
P4は、C2とXLの良いとこ取り、で、C2の精度を保ちつつXLの長さを獲得した酵素です。
例えば、4.65Mbpのゲノムサイズのバクテリアを、C2、XL、P4の3種類で読んだデータがPacBioのプレゼンにあります。
10kbライブラリでセルを2個使って読み、HGAP-Celera Assembler でアセンブリしています。
C2とP4は最終的にContigが1つになりました。
XLでContigが1つにならなかったのは何で?とツッコミたくなりますが、それには触れていません。
注目すべきはマッピングできたサブリードの精度で、
- C2 86.21%
- XL 84.09%
- P4 88.28%
さらにContigの精度も、Phredスコアで言うと
- C2 58 (21x, 97x)
- XL 49 (16x, 113x)
- P4 57 (22x, 112x)
括弧内最初は、アセンブルする直前の、HGAPでエラー補正した後のリードカバレッジ、次はHGAPエラー補正する前のリードカバレッジ
つまりP4では、セル2個使って112カバレッジのリードを得、HGApでエラー補正して22xに減ったものの、22xを使ってアセンブルしたらQV57のContigが1本できた、という意味です。
私たちでも、20kbライブラリを作って、P4酵素で読んでいます。
結果は素晴らしいものでした。
これは日を改めて、お知らせしたいと思います。
「NGS現場の会」のネタにもしたいと思っています!!
2013年6月10日月曜日
メチル化検出の限界とチャレンジ
最近良く聞かれる質問をシェアします。
PacBioによるメチル化検出のことです。
PacBioのSMRTシークエンスと言えば、昨年から、メチル化をダイレクトに読める、という触れ込みで宣伝してきました。
本社のホームページでも、メチル化(正確にはBaseModification=塩基修飾)をBisulfite無しで検出できる!というのを、Pacならではの技術だと宣伝しています。
http://www.pacificbiosciences.com/applications/base_modification/
(余談ですが、このPacBioのページ、セキュリティの関係か、見れないこともあるようです。特に大学内から見ようとするとページが見つかりませんエラーが出ることもあります。)
論文等で良く出てくるのが、バクテリアの塩基修飾の検出です。 これはもう、ほぼ確立された技術と言っても良いでしょう。
昨年の論文になるけれども例えば、
1. Fang et al. Genome-wide mapping of methylated adenine residues in pathogenic Escherichia coli using single-molecule real-time sequencing, Nature Biotechnology (2012)
2. Schadt et al. Modeling kinetic rate variation in third generation DNA sequencing data to detect putative modifications to DNA bases, Genome Res. (2012)
3. Murray et al. The Methylomes of Six Bacteria, Nucl. Acids Res. (2012)
バクテリアのメチル化で有名(?)なのに 5-メチルシトシン、4-メチルシトシン、6-メチルアデニン、があります。
以前、ここのブログ(Base Modification 塩基修飾 (新)で、検出のされ方について書きました。
なので、IPDとかの説明は省略しますが、上の図の右側が、IPD-Ratioのシグナチャーです。
4mCと6mAは、はっきりとピークがあることがわかりますね。
ところが5mCに関しては、「えっ、どこ?」というくらいわかりにくい。
そこで、Tet1という酵素の登場です。
Tetは5mCを化学反応で5‐カルボキシシトシンに変えてしまうのです。
5caCに変換してからPacで読んでみると、このとおり(下図の右)、IPD-ratioがはっきりとしてくる!
と、まあここまでは、バクテリアの塩基修飾検出、では良いのですが、相手が真核生物となると、新たな問題(というか挑戦)が出てくる。
「5-メチルシトシンと、5‐ハイドロキシメチルシトシンは見分けつくのか?」
今のところ、できるという保証はできません。 否、できるかもしれません。 できたらそれで一本書けるでしょうね。 (と、だんだん表現を肯定的に変更)
なぜかというと、Tetでの変換はそもそも、5mCを5hmCに変換し、その後いろいろステップがあって最終的に5caCになります。
つまり、5mCは全て、5hmCに変わってしまうので、5caCになったあとで、どれが5mC由来でどれが5hmC由来だったかわからない!
というわけなんですが、理論的には、5hmCをグリコシル化しTet変換から守ることで、5mCだけを5hmCと区別して検出することはできそうです。
できそう、と書いたのは、やったことが無いので、本当に出来るか自信が持てないから。
PacBioで試しているかどうかはわかりません。
もしかすると、ユーザーの誰かが挑戦しているかな?
高等真核生物では、メチル化と言っても、ゲノムの色々な場所に集まって存在するケースが多いですよね。
CpGアイランドとかインプリンティング領域とか。
そういうところでは、メチル化のIPD-Ratioのピークがマージされて、別のシグナチャーになってしまいます。
ゲノムに比較的バラバラに存在するバクテリアの時と、同じアルゴリズムでは検出できないので、そこを今、PacBioでは時間をかけて開発しています。
2013年6月5日水曜日
アドバンストワークショップ in TOKYO
夏ですねー、この天気。
梅雨入りしたのに東京は晴天で暑い!
先日ここで宣伝させて頂いた5月24日半日セミナーは満員御礼の大反響でした!
ランチが足りなく急きょピザを注文したりして、皆さまご協力ありがとうございました。
お客様のアンケートにも、「またこういうセミナーを開いて欲しい」「今度はトランスクリプトームのテーマが良い」など、とても前向きなご意見ご感想があり、嬉しい限り。
今度はいつ企画しようかな?
イルミナさんがやっているような、ウェビナーも良いですよね。 公開OKな方がいらっしゃればそれもまた選択肢のひとつ。
ところで、今週、PacBioのユーザー様を対象にした、ワークショップを行っています。
このワークショップは、ウェットを中心とした、PacBio最新プロトコルを学んで頂くことを目的としています。
最大の見せ場?は、20kbの超ロングライブラリーを作成して読む!
あと、今までのSMRT Bellライブラリを作らない方法で、読む!
皆さま一生懸命ライブラリ作成中です。
このワークショップには、PacBio本社からアプリのトレーナーを1名呼び、トミーデジタルのアプリ担当者3名、PacBioアジアオフィスからの社員1名と、さらにさらに、ライブラリーサイズセレクションの機器「Blue Pippin」の販売元本社と日本代理店(日本ジェネティクス株式会社)から計3名が、サポートに参加しています。
ありがとうございます。
私は今回は脇役で。
データが出てくるのは金曜くらいですが、皆さん楽しみですね。
ワークショップの様子(Blue Pippinの操作を見学しているところ)
ワークショップ会場は、上野動物園の隣りの、池之端のエッジビルというところです。
梅雨入りしたのに東京は晴天で暑い!
先日ここで宣伝させて頂いた5月24日半日セミナーは満員御礼の大反響でした!
ランチが足りなく急きょピザを注文したりして、皆さまご協力ありがとうございました。
お客様のアンケートにも、「またこういうセミナーを開いて欲しい」「今度はトランスクリプトームのテーマが良い」など、とても前向きなご意見ご感想があり、嬉しい限り。
今度はいつ企画しようかな?
イルミナさんがやっているような、ウェビナーも良いですよね。 公開OKな方がいらっしゃればそれもまた選択肢のひとつ。
ところで、今週、PacBioのユーザー様を対象にした、ワークショップを行っています。
このワークショップは、ウェットを中心とした、PacBio最新プロトコルを学んで頂くことを目的としています。
最大の見せ場?は、20kbの超ロングライブラリーを作成して読む!
あと、今までのSMRT Bellライブラリを作らない方法で、読む!
皆さま一生懸命ライブラリ作成中です。
このワークショップには、PacBio本社からアプリのトレーナーを1名呼び、トミーデジタルのアプリ担当者3名、PacBioアジアオフィスからの社員1名と、さらにさらに、ライブラリーサイズセレクションの機器「Blue Pippin」の販売元本社と日本代理店(日本ジェネティクス株式会社)から計3名が、サポートに参加しています。
ありがとうございます。
私は今回は脇役で。
データが出てくるのは金曜くらいですが、皆さん楽しみですね。
ワークショップの様子(Blue Pippinの操作を見学しているところ)
ワークショップ会場は、上野動物園の隣りの、池之端のエッジビルというところです。
2013年5月15日水曜日
HGApの論文のデータについて
Nature Method からHGApの論文が出ました。 (http://www.ncbi.nlm.nih.gov/pubmed/23644548)
PacBioの社運を賭けて?では無いでしょうが、昨年から相当力を入れて開発していましたよ。
そこで、この論文の話・・・と行きたいところですが、今日はそうではなく、気になったことがあるのでお知らせします。 元データの話です。
最初に気づいたのは私ではなくCLC-Bio Japanの宮本さんで、彼女が教えてくれたところによると、EBIで落とせるfastqは、リード数が極端に少ないそうです。
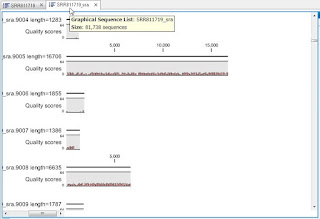
本論文中に出てくる、大腸菌の1セル分のデータ、例えばSRR811719
http://www.ebi.ac.uk/ena/data/view/SRR811719
は、 EBIのサイトでは、リード数 81,741 と書いてあるんですが、落としてみると 297本しかない! (宮本さんから教えて頂いたのでGenomics Work Bench で表示します! GWBが297本にフィルタリングしたわけでは無いので念のため)
というわけで、EBIにアップされているこのPacの配列は使えませんね。どこか間違っているのかな?
そうなってくると気になるのは別のデータベース。
NCBIのSRAではどうか?
同じデータSRR811719を見てみます。(http://www.ncbi.nlm.nih.gov/sra/?term=SRR811719)
こちらは.sra フォーマットで出来ているので、fastqフォーマットに変換するには、sratoolkitというツールをNCBIから落としてインストールし、
fastq-dump SRR811719.sra -A SRR811719
という感じでfastqフォーマットに変換します。
すると、
おおーっ! 81,738本になった!
これでも実はまだ使えません。
なぜかというと、この本数は、ベースコールデータとして使えないリードを含む、全てのリードの本数だからです。
この論文のデータが出された時は、1回に読めるZMWの数は75,000です。
なのに81,738本というのは、明らかに、読まれるウェルの数を超えています。
この差分の正体は、主に、データ補正用のウェル、です。
本当にデータが出てくる予定のウェルは75,153個です。
しかし、そのうち信頼できるデータが出てくるウェルは大体2割から3割。
なので、この8万本のリードから、本当にデータとして使えるリードは、15,000~22,000本なのです。
さらに、上記のグラフをよく見ると、15k以上のリードもある。
つまり(ライブラリー)‐(アダプター)‐(ライブラリー)という配列を含んでいるので、アダプター配列は邪魔。
本当はライブラリーだけの配列=サブリードだけの配列が良いのですが・・・。
我がDDBJも、sraフォーマットだったので、SRAのと同じではないか、と勝手に判断しました。 すみません。
さ ・ ら ・ に !
論文を見ると、リード長500、Read Quality 80 で最初にフィルタリングして、そのサブリードを、アセンブリ解析に用いています。
こうなってくると、PacBioが出しているSMRT Analysis というフリーの二次解析ソフトが必要になってきます。 (そんなん無くても自力でツールを作れる方もいるでしょうが)
ちょっと、Pacのデータってどんなもんかなー、的に見てみたいひとには敷居が高く感じてしまいますね。
同じデータ、しかしPacBioRS出力の本当の生データ、は誰でもPacのサイトからダウンロードできます。 https://github.com/PacificBiosciences/DevNet/wiki/Datasets
"E. coli, M. ruber and P. heparinus with assemblies using HGAP beta implementation"
フォーマットはbas.h5 ファイルですのでちょっと普通のNGSソフトでは扱いにくいですね。
そこで、この論文の生データを、SMRT Analysisを使って論文と同じフィルタリング条件でフィルターしたのち、fastqフォーマットに変換したので、欲しいひとには差し上げようかと思います。
欲しいひといるかな?
大腸菌の例で言うと、8セルあって、フィルタリング後の塩基数が464,366,080bp
論文より若干多いですが、そこはRQフィルタリング時の誤差でしょうか。
でもリード長の分布は論文のものに似ているので、良いでしょう。
HGApも流しているので、その結果が楽しみです。
登録:
投稿 (Atom)