知らなかったのですが、こんなのがあるそうです。
日本にある、論文にはなっていないけれどお助けツールとして広く知られている、役に立っている、(基本無料で公開している)ライフサイエンス関連の情報についてのグランプリ賞です。
データベース部門、ソフトウェア部門、ウェブ部門、とあって、恥ずかしながら自分でも知らないものもありました。
そこに私のブログが2つ、ノミネートしているのを発見! おおっ!
投票締め切りは9月27日、急げ!
それはさておき、「NGS現場の会」でお話しした、最近のPacBioのアプリケーションを紹介します。
まずは「パンドラウイルス」
今年の7月に公開された、ゲノムサイズがウイルスのくせに2.5MBもある巨大ウイルスの、ゲノム解読に、PacBioが使われました。
論文になっています。 解析の詳細は、サプリメントに書いてありますので、一読をお勧めします。
もっとも、PacBioだけではなく、Illuminaと454でも読んでいて、それぞれでContigを作ったあと、いったんPhrapでマージし、その後、PacBioのロングリードでScaffoldを作ったり、Gapを埋めたり、しています。
面白いのが最後に、ContigをIlluminaデータでエラーコレクションしていること。
エラーコレクションと言えば、アセンブリする前にすることしか私の頭になかったのですが、ScaffoldingにPacBioリードを使うのであれば、後でコレクションするのもアリなんですよね。
続いて、「100K Foodborne Pathogen Genome Project 」
去年から行われているこのゲノムプロジェクトは、食中毒原因菌を100,000種類(株)読んで、ゲノム配列やメチレーションのデータを公開しています。
サルモネラ菌、リステリア菌、大腸菌、など5種あまりを優先的に読んでいて、今後はもっと広げていく予定です。
このプロジェクトでは、PacBioだけで読んだりしているゲノムがあります。
2013年7月に、20種類のゲノム解読を完了して公開しました。
最近の情報では今年の秋に、1,500種類を完了して公開するそうです。
ちなみに、このプロジェクトには、PacBioのほかにもいくつかの企業が参加していて、CLC-BIOの名前もありました。
続いて「ハイブリッドHGApによる大型ゲノムアセンブリへの応用」
ゲノムサイズが350MBあるアブラムシゲノムアセンブリに、PacBioのリードを使ってHGApしたのですが、ユニークなのが、最初のPre-Assemblyに、PacBioロングリードだけではなく、IlluminaでアセンブリしたContigをも用いていることなんです。
最新バージョンのHGApでは、PacBioロングリードだけではなくて、任意のFASTQファイルも、Pre-Assembly(エラーコレクションステップ)に使えるのです。
イリノイ州立大学のチームによるこの結果は、間もなく論文になるでしょう。
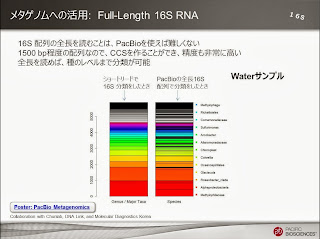
「完全長の16S配列」も、PacBioロングリードならではのアドバンテージが生かせる分野です。
1500bpくらいの配列なので、CCSでも十分よめる長さです。
韓国のチームによって出された結果は、今年のアメリカでの微生物学会にてポスター発表が行われました。そのポスターは再配布可能(というか「宣伝して」と言われている)なので、もし欲しい方がいらっしゃればお知らせください。






0 件のコメント:
コメントを投稿