先月の国際ゲノム会議で、トミーデジタルの企業セミナーを聞いた方は、「あれっ? PacBioじゃないの?」って思ったでしょう。
もちろんその後のアカデミックセッションで、PacBioのCSO, Jonas Korlach氏の講演があったので安心したのではないでしょうか。
PacBioの話が続くのはアレなんで、うちのセミナーは別の製品、それもショートリード用のライブラリ作製キット、という驚きのセミナー!
「でもそんな、今更ライブラリ作製キットなんて、売れるの? 競合たくさんあるじゃん」
と思った方、そのとおり。 競合だらけです。
でも、この製品はきっとニーズがあるだろう、と思うのはそのスタート量の少なさ。
Swift Biosciences社、(スイフト・バイオサイエンシズと発音)、愛称は「スイちゃん」
このキットは4つあり、どれも少量DNAからスタートしてライブラリを作製できるところが特徴です。
少量DNAといってもどれくらいの量からスタートできるのか?
キットの種類、PCRの有無でも異なりますが、例えば2本鎖DNAスタートなら、PCR有りで10pgから、PCR無しで10ngからライブラリを作ることができる!
そんな少量からライブラリを作りたいというニーズはあるのか?
例えばcfDNA (セルフリーDNA)。 血中に漂うDNAを回収してシークエンスしたいというニーズはあるそうです。
リキッドバイオプシーとも呼ばれます。
厳密には、対象疾患によってその用語の使い分けがされているそうですがここではどちらも、cfDNAと呼ぶことにします。
そういうcfDNA、微量DNAからのショートリード用ライブラリ作製には、スイちゃんが向いています。
先ずは2本鎖DNAから
これはライブラリ作製の段階で、5’側にアダプターを付ける反応と、3’側にアダプターを付ける反応を別々に行なっています。
どうやってそんなことができるのか? 残念ながらそれはまだ企業秘密です。
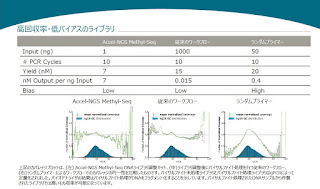
反応ごとの回収率が非常に高く(>90%)、そのため少量DNAからスタートしても、シークエンスに必要な十分量なライブラリができるのです。
続いて、1本鎖DNAからのライブラリ調製キット
1本鎖、と銘打っていますが、損傷が多い2本鎖でも応用できます。
最初にDenatureのステップがあり、全て1本鎖にしますので。
まず、3’側にアダプターを付け、相補鎖を伸ばします。
反対側にもアダプターをつけて、PCR増幅して完成。
DNAインプット量は、PCRありだと10pgからスタートできるそうです。
続いてメチレーション用キット
BiSulfiteしてから、ライブラリをつくります。
損傷してニックが入ったDNAは、それぞれ分けて、ライブラリをつくります。
なので、先にライブラリを作ってからBisulする方法に比べて、DNAを無駄にすることが無い。
インプットDNAの最低量はなんと100pgから!
どれもこれも、本当?という数字ですよね。
実は私も???
アメリカの本社が出している数字ですし、それなりにデータもあるようなので、一応信じています。
販売されて間もないので、アメリカでもケーススタディのようなものはまだありません。
まさに、今なら、アメリカのユーザとタイムラグほとんど無しで、使用できる!!
私はこれが一番のメリットだと思うんです。
研究者にとってはある程度リスクだと思いますが、最新のテクノロジーはまず試してみて、良かったら使い続ける。
既にゲノム会議でも多くの興味を頂いているので、そのうち日本で、これが本当に「使える」のかどうか、わかる日も近いです。
「スイちゃん使える!」とわかったら一気に広まりそう、というのは甘いかな?
既に広く使われている、カッパくんがいるからねえ。
手間は多分、スイちゃんの方が簡単。
あとは、少量DNAからスタートして作られた、ライブラリのクオリティとシークエンスの結果。
ここで、「カッパくんよりスイちゃんのほうがすごい。コスパも良い」ということになれば・・・
これ、PacBioのライブラリにも使える?
という質問には、「うーん、まだちょっと難しいかな。でも不可能ではないし、目的によっては、応用できるかも」とお答えしましょう。
今のところ、Illuminaシークエンサー、1本鎖用はIllumina&Ion Torrentシークエンサー用に作られています。
試薬なので、詳しくは
こちらのページまで