Nature Method からHGApの論文が出ました。 (http://www.ncbi.nlm.nih.gov/pubmed/23644548)
PacBioの社運を賭けて?では無いでしょうが、昨年から相当力を入れて開発していましたよ。
そこで、この論文の話・・・と行きたいところですが、今日はそうではなく、気になったことがあるのでお知らせします。 元データの話です。
最初に気づいたのは私ではなくCLC-Bio Japanの宮本さんで、彼女が教えてくれたところによると、EBIで落とせるfastqは、リード数が極端に少ないそうです。
本論文中に出てくる、大腸菌の1セル分のデータ、例えばSRR811719
http://www.ebi.ac.uk/ena/data/view/SRR811719
は、 EBIのサイトでは、リード数 81,741 と書いてあるんですが、落としてみると 297本しかない! (宮本さんから教えて頂いたのでGenomics Work Bench で表示します! GWBが297本にフィルタリングしたわけでは無いので念のため)
というわけで、EBIにアップされているこのPacの配列は使えませんね。どこか間違っているのかな?
そうなってくると気になるのは別のデータベース。
NCBIのSRAではどうか?
同じデータSRR811719を見てみます。(http://www.ncbi.nlm.nih.gov/sra/?term=SRR811719)
こちらは.sra フォーマットで出来ているので、fastqフォーマットに変換するには、sratoolkitというツールをNCBIから落としてインストールし、
fastq-dump SRR811719.sra -A SRR811719
という感じでfastqフォーマットに変換します。
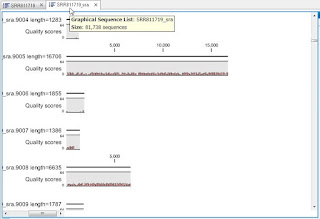
すると、
おおーっ! 81,738本になった!
これでも実はまだ使えません。
なぜかというと、この本数は、ベースコールデータとして使えないリードを含む、全てのリードの本数だからです。
この論文のデータが出された時は、1回に読めるZMWの数は75,000です。
なのに81,738本というのは、明らかに、読まれるウェルの数を超えています。
この差分の正体は、主に、データ補正用のウェル、です。
本当にデータが出てくる予定のウェルは75,153個です。
しかし、そのうち信頼できるデータが出てくるウェルは大体2割から3割。
なので、この8万本のリードから、本当にデータとして使えるリードは、15,000~22,000本なのです。
さらに、上記のグラフをよく見ると、15k以上のリードもある。
つまり(ライブラリー)‐(アダプター)‐(ライブラリー)という配列を含んでいるので、アダプター配列は邪魔。
本当はライブラリーだけの配列=サブリードだけの配列が良いのですが・・・。
我がDDBJも、sraフォーマットだったので、SRAのと同じではないか、と勝手に判断しました。 すみません。
さ ・ ら ・ に !
論文を見ると、リード長500、Read Quality 80 で最初にフィルタリングして、そのサブリードを、アセンブリ解析に用いています。
こうなってくると、PacBioが出しているSMRT Analysis というフリーの二次解析ソフトが必要になってきます。 (そんなん無くても自力でツールを作れる方もいるでしょうが)
ちょっと、Pacのデータってどんなもんかなー、的に見てみたいひとには敷居が高く感じてしまいますね。
同じデータ、しかしPacBioRS出力の本当の生データ、は誰でもPacのサイトからダウンロードできます。 https://github.com/PacificBiosciences/DevNet/wiki/Datasets
"E. coli, M. ruber and P. heparinus with assemblies using HGAP beta implementation"
フォーマットはbas.h5 ファイルですのでちょっと普通のNGSソフトでは扱いにくいですね。
そこで、この論文の生データを、SMRT Analysisを使って論文と同じフィルタリング条件でフィルターしたのち、fastqフォーマットに変換したので、欲しいひとには差し上げようかと思います。
欲しいひといるかな?
大腸菌の例で言うと、8セルあって、フィルタリング後の塩基数が464,366,080bp
論文より若干多いですが、そこはRQフィルタリング時の誤差でしょうか。
でもリード長の分布は論文のものに似ているので、良いでしょう。
HGApも流しているので、その結果が楽しみです。