って驚いたかもしれません。
ハイ、この論文、昨年末に出ました。
Coupland P. et al. (2012) Direct sequencing of small genomes on the Pacific Biosciences RS without library preparation. Biotechniques 53, 365.
PubMed
PacBioのシーケンスのハードルは、最初に用意するDNA量が多いことです。
Shearing前には5μg以上必要なときもあります。
そこでこのスターティング量を減らすべく、MagBeadsという磁気ビーズでロードする方法が開発されたのですが、さらにさらに少ない量しか無い時の、プロトコルの話です。
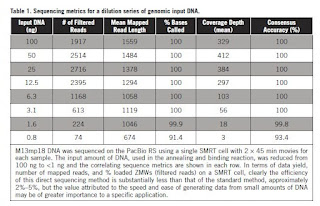
詳細は、割愛するとして、まずは出力結果と精度から。
上記の論文より引用
100ngから読むことができ、最少では0.8ngのInput DNA 量というのは驚きです。
ただし、リード数は少ないです。
ですが、少ないInput量でもちゃんと読める(コンセンサス精度が100%)、ということと、Wellcome Trust Sanger Instituteという、権威ある、Pac以外の第三者が出している論文ということで社内でも評価は高いです。
Pacのソフトでもちゃんと解析してくれて、Base Modificationまで検出できた、などと言ってくれるのは嬉しい!
さて、このような、SMRT Bell無しでのシーケンスへの取り組みは、かなり前から行われています。
すでにいくつかのプロトコルが試されていて、少量のDNAからの読み取りに成功していると聞きます。
今はダンベル型のライブラリですが、近い将来、線形ライブラリも出てくるかもしれません。
約束はできませんけれど。
当たり前ですが、線形ライブラリになった場合、Circular Consensus はできませんね。
10kbとか、長いインサート用になるでしょう。
長いインサートで、かつインプット量が少なくて済むなら、・・・ うーん、需要は高そうです。
10x 20x取れれば、コンセンサスの精度はQV50(99.999%)以上になるので、例えば、forensicsみたいにごく少量のDNAしか取れないような分野に応用できないかあ、と思ったり。

0 件のコメント:
コメントを投稿