「アベノミクス」、「今でしょう?」、この2つのどっちかじゃないか、と今から予想しています。
http://singo.jiyu.co.jp/index.htmlを見ると、ああ、そういう言葉もはやったね、と思いますが、流行語ってほとんど定着しないんですね。
アベノミクスが来年忘れられるようでは困りますが、「今でしょう?」はまさに今しか受けない、というか、そろそろ使用期限が切れるフレーズかな?
小学生の間ではまだすごいブームらしいです。
さてさて、ユーザーの方にはもちろんアナウンス済みですが、PacBioはハード・ソフトの両面でバージョンアップします(しました)。
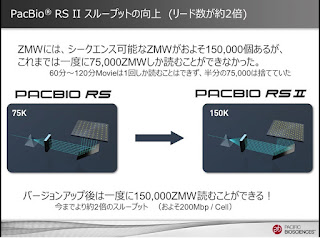
ハードは、以前「第一回アジアユーザーミーティング in シンガポール」でも触れた、RSIIです。
これは今まで、一度に読めるZMWの数が75,000だったのが、レーザーの光を分散させる光学機器の改善で、一度に150,000読めるようになったことです。
120分Movieで今まで750,000しか読めなかったのが2倍読めるようになった。つまりデータスループットが2倍になったことを意味します。
1セルあたりおよそ200Mbp、というのは控えめかもしれません。
私たちがあるバクテリアを10kbライブラリで読んでみた時、Productivity=1(信頼のあるリードデータ)が31%くらいのデータで、220Mbpでした。
その時はN=3でしたが、以来サンプルを変えて何セル読んでも大体今までの2倍は読める。
宣伝に偽り無し、でほっと一安心! (← ユーザーの立場での感想)
上図の、左側が今までのRS、右がRSII
リード長がX軸で、Y軸にその本数を示しています。 縮尺を合わせました。 どちらもProductivity=1が31%で、RSが102Mbp、RSIIが221Mbpのスループットでした。
分布「形」にはあまり差は無いのがわかるでしょう? 高さは大きく変わっています。
バージョンアップのソフトの方、こちらは、 酵素です。
P4という酵素がリリースされました。
こちらは、すごい改善! というわけでは無いのですが、より安定して、今までと同じく長く読める酵素です。
今まで、C2とXLという2種類の酵素がありました。
C2は精度は良いがスピードが遅いので長さがいまいち(平均3300bpくらい)
XLは精度が若干落ちるがスピードが速いので平均4000bpくらい
P4は、C2とXLの良いとこ取り、で、C2の精度を保ちつつXLの長さを獲得した酵素です。
例えば、4.65Mbpのゲノムサイズのバクテリアを、C2、XL、P4の3種類で読んだデータがPacBioのプレゼンにあります。
10kbライブラリでセルを2個使って読み、HGAP-Celera Assembler でアセンブリしています。
C2とP4は最終的にContigが1つになりました。
XLでContigが1つにならなかったのは何で?とツッコミたくなりますが、それには触れていません。
注目すべきはマッピングできたサブリードの精度で、
- C2 86.21%
- XL 84.09%
- P4 88.28%
さらにContigの精度も、Phredスコアで言うと
- C2 58 (21x, 97x)
- XL 49 (16x, 113x)
- P4 57 (22x, 112x)
括弧内最初は、アセンブルする直前の、HGAPでエラー補正した後のリードカバレッジ、次はHGAPエラー補正する前のリードカバレッジ
つまりP4では、セル2個使って112カバレッジのリードを得、HGApでエラー補正して22xに減ったものの、22xを使ってアセンブルしたらQV57のContigが1本できた、という意味です。
私たちでも、20kbライブラリを作って、P4酵素で読んでいます。
結果は素晴らしいものでした。
これは日を改めて、お知らせしたいと思います。
「NGS現場の会」のネタにもしたいと思っています!!