今日は久しぶりに会社のチームみんなで集まってビアガーデンパーティをしました。
出張が多いメンバーが一度にみんなそろうことはめったに無いのです。
9月30日、ビアガーデンとしてはちょっと肌寒い時期でしたが、大手町サンケイビル4階の「天空のビアガーデン」
今年の夏を締めくくるにふさわしい、とても良いひと時でした。
さてさて、バクテリアサイズのゲノムのデノボアセンブリには、PacBio RSを超える機械はこの世に存在しない! というのはこのブログを読んでいる皆様にはもう常識?ですが、海外でももちろんその通りです。
JGI(Joint Genome Institute)という、アメリカエネルギー省管轄の研究所がカリフォルニア州ウォルナットクリークにありまして、ここでは2台のPacBio RSがフル活動しています。
エネルギー省の施設なので、読んでいるバクテリアの種類は、病原菌というよりもどちらかというと環境や生態系に関する重要バクテリアや、バイオ燃料開発などで重要なバクテリアで、これらをがんがん読んでいるそうです。
彼らのホームページにもPacBioシークエンサーのことが書かれていて、HGApのことについても触れられています。HGApはもうご存知、PacBioのロングリードだけでエラー補正からアセンブリ、Contig Polishingまでを自動で行うパイプラインのことですね。
このアルゴリズムの開発にも、JGIは大きな貢献をしました。
http://www.jgi.doe.gov/News/news_13_05_06.html
さて、JGIでは、大量のサンプルをガンガン読んでいるために、サンプル調整を手で行うのは大変になりました。
そこで導入したのが、PerkinElmer社から売られている自動分注装置。
Caliper社のScicloneという自動サンプル調整ロボット機械には、PacBio用のアプリケーションが作られ、その名も Maestro PacBio 10Kb DNA Library Prep
マエストロですよ!好きです、こういう自身満々の名前
http://www.perkinelmer.com/Catalog/Family/ID/Sciclone%20NGS%20Workstation
この装置は、PacBioの3Kb - 10Kbのライブラリ調整を自動で行ってくれるそうです。
スループットは一度に8 から 96 サンプルで、8時間以内で終了するそうです。
このような装置は、いろんな種類のサンプルを読む場合、たくさんのライブラリを作る必要があるので、大活躍するかもしれませんね。
反対に、大型ゲノムを数種類だけ読むような場合、最初にライブラリを作ってしまえばあとはできているライブラリをたくさん読むだけなので、サンプル調整は手で行った方が安上りかもしれません。
いずれにせよ、たくさんの種類のバクテリアを読んでいるJGIには、この手の機械が有用なのでしょう。
これがその自動化マシンを使ったときの、DNAサンプルからContigデータまでの流れです。
PacBioのプレゼンより引用
DNAのシェアリングまではG-tubeでやるんですね。ここはマニュアルかあ。
断片化した後の、ライブラリ調整を、PacBioプロトコルに合わせてこの装置がやってくれるわけですね。
出来上がったライブラリをシークエンサーに乗せるわけですが、サイズセレクションは当然これもマニュアルでしょうね。
回収率はどれくらいなんでしょう?
2013年9月30日月曜日
2013年9月29日日曜日
公式アナウンスメント: Roche Diagnostics と協力して診断機械開発へ
http://investor.pacificbiosciences.com/releasedetail.cfm?ReleaseID=793199
この発表はご覧になった方もいるかと思います。
Roche Diagnosticsと言えばみなさんご存じ、454 GS FLX、GS Junor のメーカーですね。
スイスに本社を置くRoche社は、2012年現在全世界で82,000人の社員を抱え、R&Dに80億スイスフラン(約8672億円)を投資し、455億スイスフラン(約4兆9322億円)を売り上げる、世界最大のバイオテックカンパニーです。
診断の分野でも大きなシェアを持っているそうです。
そんなRocheとPacific Biosciencesが協力して、ヒトIn vitro診断機器を開発する、というアグリーメントを結びました。
おそらく、噂が立つと思いますので先に、これだけはお伝えしておきます。
RocheがPacBioを買収するという話ではありません!!
アグリーメントの内容は、このようなものです。
- PacBioは、ヒト in vitro 診断のための新しい機械をRocheのために開発する
- 新しい機械は、PacBioのSMRTテクノロジーを使用する
- Rocheはこの新しい機械を全世界で独占販売する権利を有する
- 開発資金として、3500万ドルをRocheから受け取り、さらに4000万ドルを受けとる予定である
- PacBioは、ヒト in vitro 診断以外の分野では、これまでどおり機械の開発・販売を続ける
さっそくいろんな噂がツィートされているみたいです。
でも、噂の類はここには書きませんので、ご安心ください。
2013年9月25日水曜日
アメリカユーザーグループミーティングin Palo Alto
たまにチェックするブログがあります。 (http://www.homolog.us/blogs/blog/2013/09/21/end-illumina-era)
ここに、先週、カリフォルニア州パロアルトで行われた「北米・PacBioユーザーグループミーティング」の内容が書かれています!
私も今回は参加したのでわかりますが、このブログはすごく良くまとまっているので一見の価値ありです!
「ショートリードの時代の終わり?」と題していますが、バクテリアデノボの世界ではまさにそうでしょう。
今回のグループミーティングでも、バクテリア関連(デノボアセンブリ、メチレーション)が4割、トランスクリプトームが3割、高等生物アセンブリが2割、MHCなどその他が1割、という割合だった気がします。
およそ60人が参加した今回のグループミーティング(休憩時間とランチタイムの様子)
さて、私が興味を持ったトピックスは、バクテリア以外です。
1.Arabidopsis(Salk Institute for Biological Studies);Arabidopsisは、Colombia 10のゲノム配列が読まれ公開されていますが、まだ800株ほどの読まれていない株があるそうです。
彼らはPacBioのリードだけを用いてアラビの株をたくさん読んで、株間のゲノム比較を行っています。
Ler-0 株を例にとると、20kbライブラリを作り、ゲノムサイズ120Mbの50x分のデータを得ました。
HGApを使ってアセンブリしたところ、Contig本数は540本、塩基総数は124Mbp、N50 = 6.2Mbp、Max 13Mbpという結果を得ました。
この、20kbライブラリ作製のところで、BluePippinを使っています。
Col0株も読んでおり、TAIR10のリファレンスゲノムとの比較では99%以上の一致を得たので、Ler-0株もそれなりに高い精度のContigが作られているという感触を持っているみたいです。
現在多くのSNPやInDelが見つかっているのでそれらを検証しているとのこと。
1株のアセンブルに要した時間はおよそ10日。うち9日間はHGApのPre-Assembly(最初のエラーコレクション)に要した時間です。 Pre-Assemblyの時間を短くするのが課題ですね。
実はSMRT Analysisの次のバージョンで、Pre-Assemblyが劇的に速くなるオプションがつくのですが、まだベータ版です。
2.チキンRNA-Seq(Gladstone Institute);ニワトリの心臓とヒトの心臓は構造が似ているため、モデルにはちょうど良いそうです。 しかしニワトリのRefSeq情報はまだ6,500配列しかなく、不十分。 そこで完全長cDNA=Isoformの配列データを得る必要があるという。
今までショートリードで読み、Trinityなどのツールを使って解析してきたが、今回PacBioを使ってIsoformデータを得ることに挑戦したそうです。
0-1kb,1-2kb,2-3kb,>3kbの4種類のライブラリを作り、フルパスサブリード配列のみを対象に、イルミナのリードでエラーコレクションし、ゲノム参照配列にアラインしました。 Geneアノテーションを行ったところ、新しいTranscriptが2000程見つかったそうです。
3.ヒトTranscriptome(Stanford Univ);LSCという配列圧縮/エラー補正アルゴリズムを用いて、完全長cDNAの解析を行っていました。スプライシングの検出には完全長が不可欠です。 曰く、Cufflinksなどのショートリード向けのツールは、ベースにしている理論が数学的でありバイオロジカルな現象を反映して無い、と。
エラーコレクションを行った後のPacBioリードを、BLATでゲノムにアラインしました。 8,084本の既知の転写産物+5,459本の新規転写産物を得たそうです。
PacBioでは間もなく、新しい試薬がリリースされる予定です。
P5/C3 ケミストリーは、ロングリードをさらに伸ばして、平均8,500bpを実現します。
と言っても、これはポリメラーゼリード長です。 サブリード長を伸ばすには、20kbライブラリ+サイズセレクションが必須です。
そんなわけで、今回の演者で20kbライブラリを作った人はみな、BluePippinを使ったサイズセレクションを行っていました。
BPのメーカーであるSage Science社のTwitterで知ったのですが、MRSAから抽出したDNAを、20kbライブラリ+BPサイズセレクション実行後、P5/C3試薬を使い3時間Movieで読んだら、最高34,500bpのポリメラーゼリードを得たそうです!
ここに、先週、カリフォルニア州パロアルトで行われた「北米・PacBioユーザーグループミーティング」の内容が書かれています!
私も今回は参加したのでわかりますが、このブログはすごく良くまとまっているので一見の価値ありです!
「ショートリードの時代の終わり?」と題していますが、バクテリアデノボの世界ではまさにそうでしょう。
今回のグループミーティングでも、バクテリア関連(デノボアセンブリ、メチレーション)が4割、トランスクリプトームが3割、高等生物アセンブリが2割、MHCなどその他が1割、という割合だった気がします。
およそ60人が参加した今回のグループミーティング(休憩時間とランチタイムの様子)
さて、私が興味を持ったトピックスは、バクテリア以外です。
1.Arabidopsis(Salk Institute for Biological Studies);Arabidopsisは、Colombia 10のゲノム配列が読まれ公開されていますが、まだ800株ほどの読まれていない株があるそうです。
彼らはPacBioのリードだけを用いてアラビの株をたくさん読んで、株間のゲノム比較を行っています。
Ler-0 株を例にとると、20kbライブラリを作り、ゲノムサイズ120Mbの50x分のデータを得ました。
HGApを使ってアセンブリしたところ、Contig本数は540本、塩基総数は124Mbp、N50 = 6.2Mbp、Max 13Mbpという結果を得ました。
この、20kbライブラリ作製のところで、BluePippinを使っています。
Col0株も読んでおり、TAIR10のリファレンスゲノムとの比較では99%以上の一致を得たので、Ler-0株もそれなりに高い精度のContigが作られているという感触を持っているみたいです。
現在多くのSNPやInDelが見つかっているのでそれらを検証しているとのこと。
1株のアセンブルに要した時間はおよそ10日。うち9日間はHGApのPre-Assembly(最初のエラーコレクション)に要した時間です。 Pre-Assemblyの時間を短くするのが課題ですね。
実はSMRT Analysisの次のバージョンで、Pre-Assemblyが劇的に速くなるオプションがつくのですが、まだベータ版です。
2.チキンRNA-Seq(Gladstone Institute);ニワトリの心臓とヒトの心臓は構造が似ているため、モデルにはちょうど良いそうです。 しかしニワトリのRefSeq情報はまだ6,500配列しかなく、不十分。 そこで完全長cDNA=Isoformの配列データを得る必要があるという。
今までショートリードで読み、Trinityなどのツールを使って解析してきたが、今回PacBioを使ってIsoformデータを得ることに挑戦したそうです。
0-1kb,1-2kb,2-3kb,>3kbの4種類のライブラリを作り、フルパスサブリード配列のみを対象に、イルミナのリードでエラーコレクションし、ゲノム参照配列にアラインしました。 Geneアノテーションを行ったところ、新しいTranscriptが2000程見つかったそうです。
3.ヒトTranscriptome(Stanford Univ);LSCという配列圧縮/エラー補正アルゴリズムを用いて、完全長cDNAの解析を行っていました。スプライシングの検出には完全長が不可欠です。 曰く、Cufflinksなどのショートリード向けのツールは、ベースにしている理論が数学的でありバイオロジカルな現象を反映して無い、と。
エラーコレクションを行った後のPacBioリードを、BLATでゲノムにアラインしました。 8,084本の既知の転写産物+5,459本の新規転写産物を得たそうです。
PacBioでは間もなく、新しい試薬がリリースされる予定です。
P5/C3 ケミストリーは、ロングリードをさらに伸ばして、平均8,500bpを実現します。
と言っても、これはポリメラーゼリード長です。 サブリード長を伸ばすには、20kbライブラリ+サイズセレクションが必須です。
そんなわけで、今回の演者で20kbライブラリを作った人はみな、BluePippinを使ったサイズセレクションを行っていました。
BPのメーカーであるSage Science社のTwitterで知ったのですが、MRSAから抽出したDNAを、20kbライブラリ+BPサイズセレクション実行後、P5/C3試薬を使い3時間Movieで読んだら、最高34,500bpのポリメラーゼリードを得たそうです!
2013年9月24日火曜日
NGS現場の会で話したPacBio最近のアプリケーション
知らなかったのですが、こんなのがあるそうです。
日本にある、論文にはなっていないけれどお助けツールとして広く知られている、役に立っている、(基本無料で公開している)ライフサイエンス関連の情報についてのグランプリ賞です。
データベース部門、ソフトウェア部門、ウェブ部門、とあって、恥ずかしながら自分でも知らないものもありました。
そこに私のブログが2つ、ノミネートしているのを発見! おおっ!
投票締め切りは9月27日、急げ!
それはさておき、「NGS現場の会」でお話しした、最近のPacBioのアプリケーションを紹介します。
まずは「パンドラウイルス」
今年の7月に公開された、ゲノムサイズがウイルスのくせに2.5MBもある巨大ウイルスの、ゲノム解読に、PacBioが使われました。
論文になっています。 解析の詳細は、サプリメントに書いてありますので、一読をお勧めします。
もっとも、PacBioだけではなく、Illuminaと454でも読んでいて、それぞれでContigを作ったあと、いったんPhrapでマージし、その後、PacBioのロングリードでScaffoldを作ったり、Gapを埋めたり、しています。
面白いのが最後に、ContigをIlluminaデータでエラーコレクションしていること。
エラーコレクションと言えば、アセンブリする前にすることしか私の頭になかったのですが、ScaffoldingにPacBioリードを使うのであれば、後でコレクションするのもアリなんですよね。
続いて、「100K Foodborne Pathogen Genome Project 」
去年から行われているこのゲノムプロジェクトは、食中毒原因菌を100,000種類(株)読んで、ゲノム配列やメチレーションのデータを公開しています。
サルモネラ菌、リステリア菌、大腸菌、など5種あまりを優先的に読んでいて、今後はもっと広げていく予定です。
このプロジェクトでは、PacBioだけで読んだりしているゲノムがあります。
2013年7月に、20種類のゲノム解読を完了して公開しました。
最近の情報では今年の秋に、1,500種類を完了して公開するそうです。
ちなみに、このプロジェクトには、PacBioのほかにもいくつかの企業が参加していて、CLC-BIOの名前もありました。
続いて「ハイブリッドHGApによる大型ゲノムアセンブリへの応用」
ゲノムサイズが350MBあるアブラムシゲノムアセンブリに、PacBioのリードを使ってHGApしたのですが、ユニークなのが、最初のPre-Assemblyに、PacBioロングリードだけではなく、IlluminaでアセンブリしたContigをも用いていることなんです。
最新バージョンのHGApでは、PacBioロングリードだけではなくて、任意のFASTQファイルも、Pre-Assembly(エラーコレクションステップ)に使えるのです。
イリノイ州立大学のチームによるこの結果は、間もなく論文になるでしょう。
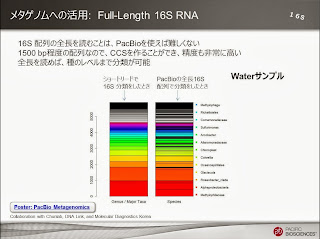
「完全長の16S配列」も、PacBioロングリードならではのアドバンテージが生かせる分野です。
1500bpくらいの配列なので、CCSでも十分よめる長さです。
韓国のチームによって出された結果は、今年のアメリカでの微生物学会にてポスター発表が行われました。そのポスターは再配布可能(というか「宣伝して」と言われている)なので、もし欲しい方がいらっしゃればお知らせください。
日本にある、論文にはなっていないけれどお助けツールとして広く知られている、役に立っている、(基本無料で公開している)ライフサイエンス関連の情報についてのグランプリ賞です。
データベース部門、ソフトウェア部門、ウェブ部門、とあって、恥ずかしながら自分でも知らないものもありました。
そこに私のブログが2つ、ノミネートしているのを発見! おおっ!
投票締め切りは9月27日、急げ!
それはさておき、「NGS現場の会」でお話しした、最近のPacBioのアプリケーションを紹介します。
まずは「パンドラウイルス」
今年の7月に公開された、ゲノムサイズがウイルスのくせに2.5MBもある巨大ウイルスの、ゲノム解読に、PacBioが使われました。
論文になっています。 解析の詳細は、サプリメントに書いてありますので、一読をお勧めします。
もっとも、PacBioだけではなく、Illuminaと454でも読んでいて、それぞれでContigを作ったあと、いったんPhrapでマージし、その後、PacBioのロングリードでScaffoldを作ったり、Gapを埋めたり、しています。
面白いのが最後に、ContigをIlluminaデータでエラーコレクションしていること。
エラーコレクションと言えば、アセンブリする前にすることしか私の頭になかったのですが、ScaffoldingにPacBioリードを使うのであれば、後でコレクションするのもアリなんですよね。
続いて、「100K Foodborne Pathogen Genome Project 」
去年から行われているこのゲノムプロジェクトは、食中毒原因菌を100,000種類(株)読んで、ゲノム配列やメチレーションのデータを公開しています。
サルモネラ菌、リステリア菌、大腸菌、など5種あまりを優先的に読んでいて、今後はもっと広げていく予定です。
このプロジェクトでは、PacBioだけで読んだりしているゲノムがあります。
2013年7月に、20種類のゲノム解読を完了して公開しました。
最近の情報では今年の秋に、1,500種類を完了して公開するそうです。
ちなみに、このプロジェクトには、PacBioのほかにもいくつかの企業が参加していて、CLC-BIOの名前もありました。
続いて「ハイブリッドHGApによる大型ゲノムアセンブリへの応用」
ゲノムサイズが350MBあるアブラムシゲノムアセンブリに、PacBioのリードを使ってHGApしたのですが、ユニークなのが、最初のPre-Assemblyに、PacBioロングリードだけではなく、IlluminaでアセンブリしたContigをも用いていることなんです。
最新バージョンのHGApでは、PacBioロングリードだけではなくて、任意のFASTQファイルも、Pre-Assembly(エラーコレクションステップ)に使えるのです。
イリノイ州立大学のチームによるこの結果は、間もなく論文になるでしょう。
「完全長の16S配列」も、PacBioロングリードならではのアドバンテージが生かせる分野です。
1500bpくらいの配列なので、CCSでも十分よめる長さです。
韓国のチームによって出された結果は、今年のアメリカでの微生物学会にてポスター発表が行われました。そのポスターは再配布可能(というか「宣伝して」と言われている)なので、もし欲しい方がいらっしゃればお知らせください。
2013年9月15日日曜日
Pacすごいぜ論文と、最新20kbサンプルデータ
Koren et.al. Reducing assembly complexity of microbial genomes with single-molecule sequencing. Genome Biology 2013, 14:R101
(http://genomebiology.com/2013/14/9/R101/abstract)
この論文は9月13日にPublishされました。
日々、PacBioのプレゼン等でこういう「Pacすごいぜ」的な情報に慣れきっている私には、それほど驚くほどのものではない、と言っては著者に失礼ですが、PacBioを知らない方やこれから学ぼうとしている方には良い参考になると思います。
ちなみに、著者は、エラー補正アルゴリズム(pacBioToCA)の作者でもあります。
さて、先々週の「NGS現場の会」の続きです。
「DeNovoの達人」で東大・笠原さんの解析フローが、公開されました。
こちら(https://cell-innovation.nig.ac.jp/wiki/tiki-index.php?page=Sprai)
デモデータで使用できるPacBioデータのリンクもありますので是非どうぞ。
E.coli 20kbのライブラリのデータを使うと、10kb以上の長さのContigは1つだけになったそうです。
詳しい手順は上記のサイトをご覧ください。
私がHGAP-CA-Quiverのフローで行ったところ、同じく1本になりました。
最初のPre-Assemblyステップで8,103bpをSeedリードに、それ以下の短いリードを補正用に使います。
そうして作られた12,418本の補正後ロングリード、総塩基数109MbpをCelera Assembly
その後QuiverでContigをPolishして得た最終Contigが、長さ4,665,426bp
その過程で作られる、サブリードの長さの分布がこちら
うーん、これはチャンピオンデータだな。
ここまで良いデータはなかなか出ないです。
10kp以上のサブリードだけでも100Mb(総塩基数)= 20X ある!
実際、20kbのライブラリをうまく作るのがキーで、サイズセレクションは必須、ということはたくさんDNAが取れることが必須、なわけですね。
2013年9月8日日曜日
「NGS現場の会」が終わって 反省(マイクの音量・声が小さかった?)
みなさんも参加されましたでしょうか?
9月4日、5日の「NGS現場の会・第三回研究会」 in 神戸
私は3日の夜に入り、いつものことながらプレゼンの練習と最終確認を深夜2時までやっていました。
ちなみに泊まったホテルは「ヴィラフォンテーヌ」という、名前は発音しずらいけれど6000円前後ですごく良い部屋&朝食が付いてくる!(http://www.hvf.jp/sannomiya/) 三ノ宮ではここおすすめ
さて、4日はいろんな発表聞いて、たくさんの方と話して、懇親会後も大勢で飲みに行って、と、現場の人間の集まりにふさわしい一日でした。
私の発表は次の日。 もちろん早起きできるはずもなく、しかし9時55分からの発表なので9時20分ごろには会場入りしました。
あとで知ったのですが、8時からのモーニングセッション「いまさら聞けない NGS超!入門」(株式会社ジナリス竹田 綾 さん)にて、このブログを紹介してくださったそうです。 ありがとう!!
私の発表は、ギャグも受けて良かった良かった。 ロードマップの話はみなさん驚いていたようで。
そしてフリーシークエンスキャンペーンの発表! <--こちら詳しくは現場の会のメーリングリストに流した後、本ブログでも紹介する予定です。
ちなみに私の話の内容は、こんな感じ
----------------------------------------------------------------------
聞き逃した方もいらっしゃると思いますので、内容についてはこのブログでも書こうと思います。
まず最初に、現在のスループットで、これだけ良い結果が出るという例を紹介します。
これは私たちが6月に、大腸菌のゲノムで20kbのライブラリを作り、それをBlue Pippin(TM)でサイズセレクションし、1セルで読んだ時のデータです。
100bp以上でフィルタリングすると平均リード長が6,149 bp
参照配列(K12-MG1655)にマッピングした時のサブリード平均長が4,968 bp
できたコンセンサスと、リファレンスとの一致は99.9967%
その時のカバレッジは86.99
このデータで示される通り、精度は高い! 99.9967%はすごいと思います。
さらに、このリードだけ(1 SMRT Cell だけのデータ)でアセンブルして、見事1本のContigができ、これの参照配列とのドットプロットはご覧の通りです。 ほとんど一緒!
私の発表の後、同じ会場で、「DeNovoの達人」という企画がありました。
これはもう、すごい!の一言。
阪大微研さんで、MiSeq、Ion Torrent、GS Jr、PacBioを使用して、同じゲノムを読んでもらい、そのデータを使って、3名の達人たちにアセンブリ解析してもらった結果を公開するという企画です。
皆さん、得意な分野で工夫して解析していました。
この企画、ツイートOKだったかどうか忘れたので、詳細は書きませんが、結論だけ言うとPacBioの大勝利でした!
もちろん、解析して頂いた東大・笠原さん(Master of DeNovo)のおかげです。
ゲノムサイズも数も知らされず、ドライだけで見事、サイズと数を、答えと一致させたのですから。
ロングリードでないと絶対つながらない配列は、ショートリードでいくら読んでもつながらないのです。
この会場、私のPacBio発表の後に、最終的にPacBioの宣伝になった「DeNovoの達人」、があったわけです。
これは偶然です。 本当に偶然です。
私は、「DeNovoの達人」の結果は、前もって知らされていませんでした。 だから結果についてはちょっと心配していたくらいです。
と、2日間、非常に充実していました。 スタッフのみなさん、お疲れ様でした。
神戸はいいところですね。
9月4日、5日の「NGS現場の会・第三回研究会」 in 神戸
私は3日の夜に入り、いつものことながらプレゼンの練習と最終確認を深夜2時までやっていました。
ちなみに泊まったホテルは「ヴィラフォンテーヌ」という、名前は発音しずらいけれど6000円前後ですごく良い部屋&朝食が付いてくる!(http://www.hvf.jp/sannomiya/) 三ノ宮ではここおすすめ
さて、4日はいろんな発表聞いて、たくさんの方と話して、懇親会後も大勢で飲みに行って、と、現場の人間の集まりにふさわしい一日でした。
私の発表は次の日。 もちろん早起きできるはずもなく、しかし9時55分からの発表なので9時20分ごろには会場入りしました。
あとで知ったのですが、8時からのモーニングセッション「いまさら聞けない NGS超!入門」(株式会社ジナリス竹田 綾 さん)にて、このブログを紹介してくださったそうです。 ありがとう!!
私の発表は、ギャグも受けて良かった良かった。 ロードマップの話はみなさん驚いていたようで。
そしてフリーシークエンスキャンペーンの発表! <--こちら詳しくは現場の会のメーリングリストに流した後、本ブログでも紹介する予定です。
ちなみに私の話の内容は、こんな感じ
----------------------------------------------------------------------
PacBioシークエンサーの基礎・特徴
PacBio Q&A
メチレーション検出の現状
サンプルプレップの全体像
塩基の損傷がシークエンスに与える影響
メチレーション検出の現状
サンプルプレップの全体像
塩基の損傷がシークエンスに与える影響
最近の注目トピックス
パンドラウイルスのゲノム解読
100K ゲノムプロジェクト
大型ゲノムアセンブリ
全長16Sシークエンス
全長HLAシークエンス
パンドラウイルスのゲノム解読
100K ゲノムプロジェクト
大型ゲノムアセンブリ
全長16Sシークエンス
全長HLAシークエンス
最後に
ロードマップとキャンペーンの紹介
-----------------------------------------------------------------------聞き逃した方もいらっしゃると思いますので、内容についてはこのブログでも書こうと思います。
まず最初に、現在のスループットで、これだけ良い結果が出るという例を紹介します。
これは私たちが6月に、大腸菌のゲノムで20kbのライブラリを作り、それをBlue Pippin(TM)でサイズセレクションし、1セルで読んだ時のデータです。
100bp以上でフィルタリングすると平均リード長が6,149 bp
参照配列(K12-MG1655)にマッピングした時のサブリード平均長が4,968 bp
できたコンセンサスと、リファレンスとの一致は99.9967%
その時のカバレッジは86.99
このデータで示される通り、精度は高い! 99.9967%はすごいと思います。
さらに、このリードだけ(1 SMRT Cell だけのデータ)でアセンブルして、見事1本のContigができ、これの参照配列とのドットプロットはご覧の通りです。 ほとんど一緒!
私の発表の後、同じ会場で、「DeNovoの達人」という企画がありました。
これはもう、すごい!の一言。
阪大微研さんで、MiSeq、Ion Torrent、GS Jr、PacBioを使用して、同じゲノムを読んでもらい、そのデータを使って、3名の達人たちにアセンブリ解析してもらった結果を公開するという企画です。
皆さん、得意な分野で工夫して解析していました。
この企画、ツイートOKだったかどうか忘れたので、詳細は書きませんが、結論だけ言うとPacBioの大勝利でした!
もちろん、解析して頂いた東大・笠原さん(Master of DeNovo)のおかげです。
ゲノムサイズも数も知らされず、ドライだけで見事、サイズと数を、答えと一致させたのですから。
ロングリードでないと絶対つながらない配列は、ショートリードでいくら読んでもつながらないのです。
この会場、私のPacBio発表の後に、最終的にPacBioの宣伝になった「DeNovoの達人」、があったわけです。
これは偶然です。 本当に偶然です。
私は、「DeNovoの達人」の結果は、前もって知らされていませんでした。 だから結果についてはちょっと心配していたくらいです。
と、2日間、非常に充実していました。 スタッフのみなさん、お疲れ様でした。
神戸はいいところですね。
登録:
コメント (Atom)