今から約3年前、2013年9月の、「Roche社から7500万ドルの出資を受けてRoche社向けの新しいシークエンサー&アッセイシステムを開発する」というニュースは、驚きと共に色んな憶測を呼びました。
その2年後、2015年10月に、Roche社向けに開発していたはずのシークエンサーが、突然公に姿を現し、またまた大きな反響を呼びました。
もちろんこれまで大きな出資を受けて開発された経緯もあり、ロッシュ社が大口のユーザになることは予想できたでしょう。
新しい機械が登場すると、いろんな予想や憶測、将来の展望や業界のシェアなど、話題が豊富になりますね。
Sequelもその通りで、これによって何が可能になるか、が、いろんなところで議論されました。
そして、今年に入り、Sequelの試薬、プロトコル、ソフトウェアのアップデートが頻繁に、まるでRSの導入時のように頻繁にありました。
ご存知のかたもいるかもしれませんが、RSは日本に入ってきたとき、C1というケミストリーからC2に変わる時期でした。
なので、それこそ毎週のようにソフトウェアのアップデートがあったりしたものです。
ユーザのかたにはご迷惑かけましたが、最先端の装置はアップデートでどんどん良くなっていく、ということでご理解いただけたかと。
実際、C2になって、平均リード長が2,000 bpへと向上!しましたし、装置もだんだん安定してきましたしね。
C2から始まり、XL-C2、XL-XL、P4-C2、P5-C3、P6-C4 と、酵素&ケミストリーがバージョンアップし、ソフトウェアもSMRT Analysis 1.xから2.3まで更新され、光学系のバージョンアップでリード数が2倍になり(RS -> RSII)、マグビーズステーションの導入で長いライブラリも効率良く読まれるようになり、ステージスタートで長いSubreadがより多く得られるようになりました。
プロトコルもたくさんつくられ、デノボシークエンス以外にも、Iso-Seqやターゲットリシーク、HLAアンプリコンシークエンスなどにも対応できるようになりました。
さ、て、
いまのSequelはどうでしょう?
Sequelについては、8月5日に、GenomeWebにて、アップデートが報じられています。
こちらは公式な発表なので、ブログに書いても良いでしょう。
(前情報として、2012年の夏から、PacBio社はベルギーのImecと共同研究を始めています。ナノフォトニクスCMOS技術についての共同開発らしいです)
Sequel用SMRT Cellの生産は、開発されたImecから、現在、大量生産できる企業に移している最中("in the midst of transitioning to a high-volume supplier")だそうです。
Cellのパフォーマンスは、量産体制が確立され次第、より向上するだろう、とのこと。
Jonas Korlach CSO は、記事の中で、アップグレードを重ねた最新のデータでは、平均5Gb/Cell (3~8 Gbのレンジ)、平均リード長は8~12kbが出ている、と言っています。
(注! インサートの長さについては述べられていません)
しかし Piper Jaffray 社のリサーチによると、ユーザからは平均2kb~6kbのリードしか出ていないそうです。
これについて、Hunkapiller CEOは、パフォーマンスが悪い原因は、Cellの供給元がまだプロトタイプサプライヤーで、量産体制でないためである、と説明しています。
パフォーマンスは今後、大幅に改善 "substantially improved" されるとのことです。
つまり、現在パフォーマンスがイマイチだという声があるが、これはまもなくCellの大量生産体制が安定すれば、大幅に改善されるということ。
今でも Korlach CSOの言うとおり、とても良いデータが出ることもあるでしょうが、安定してどのセルでも良いデータが出ることが大事ですよね。
これまでPacBioは、「次はこうなる!」って言ったことは、まあまあ実現できているので、今回もCEOのMike Hunkapillerが公式に説明していますから、私は前向きに考えます。
"will be substantially improved"
2016年8月29日月曜日
2016年8月22日月曜日
ヒトゲノムとPacBioロングリード 国内シンポジウムのお知らせ
前回のブログにも、Precision Medicineとヒトゲノムシークエンスについて書きましたが、アメリカ人類遺伝学会に行かないかたでも、東京&沖縄で、来月シンポジウムがあります。
今日はそのお知らせです。
主催はPacBio RSII のスーパーヘビーユーザーさんでもある、一般社団法人 沖縄綜合科学研究所。
9月7日(水)は那覇市、パシフィックホテル沖縄にて
9月9日(金)は東京浜松町の世界貿易センタービルにて
どちらもこちらのウェブサイトから詳細が確認でき、参加登録もできます。
日本語の発表と英語の発表が半々くらいで予定され、どれもPacBioを使った最先端の研究の発表になると思います。
台湾からのDr. Meiyeh Luは、アジアユーザーミーティングでも発表されました。エネルギッシュな女性でとにかく話すのが好きな感じでした。そのときは感染症バクテリアなどのゲノムシークエンスがテーマでしたが、今度はどうでしょう。
PacBioからはDr. Meredith H. Ashby 彼女もまた、話すのが好きな女性です。ま、女性はたいてい皆そうですかね。
Meredithとは何度かPacBioの本社で会って話したことがありますが、大学時代に日本語を取っていたそうです。でも日本に来るのは今回が初めてだそうで、楽しみにしていました。
なので今回の発表も期待できますよ。
アメリカからのゲストスピーカーは、Dr. Robert P. Sebra 彼は何度か日本にも来ていますし、私たちが主催した「PacBio現場の会1&2」でも発表してくれましたので、覚えているかたもいるのでは?
恐らく世界で一番、PacBioの装置を動かしているし経験もあるグループでしょう。
所長さんは元PacBioのCSO(最高科学責任者)で、Robertさんもまた元PacBio社員、他のラボのPIにも元PacBioがいるそうですから。
こちらの発表も期待できます。
どちらも午後1時半から夕方5時くらいまでの半日セミナーで、参加も無料だそうです。
PacBioに関する情報収集にはとても良い場だと思います。
シンポジウムやセミナーを企画して、会場手配して、演者のスケジュールを確保して、段取り決めて、って、いろいろ結構大変なんですよね。
だからこういう機会は、とっても貴重ですよ。
できるだけ事前参加登録をお願いしているようです。
と、ここまで言っておきながら、残念ながら、私は別の予定があって、参加できないんですよ。
行きたかったですが・・・。
2016年8月18日木曜日
Precision Medicine (プレシジョンメディシン) とロングリード
皆さん、Precision Medicineという言葉、聞いたことあると思います。
これが何を指すのかというと、「現在のゲノム解析技術と知をフルに活用して、個人に合ったオーダーメイド医療、予防医学も含めて、実現しよう!」というものではないでしょうか?
私の理解はそんなところです。皆さんそれぞれ少しずつ違った解釈をされているかもしれませんね。
しかーし、ここで英語の問題を1問。
「PrecisionとAccuracyの意味の違いを述べよ(PreciseとAccurateの違いでも良い)」
さあ、わかりますか?
普通に辞書引くと、どちらも正確性あるいは精度とか書いてあるかもしれません。でも、厳密には違いますよ。
答え:
Precisionとは精度。何回も測定してどれも近い数値が出た場合、精度が高いといい、再現性が高い、とも言う。
Accuracyとは正確度。測定結果が真の値に近い場合、正確度が高いといい、確度が高いとも言う。
なので、いくらPrecisionが良くても(同じゲノムを何回解析してほぼ同じSNVが測定されても)Accurateで無い場合(真の変異では無い)もある
(ググれば例が出てきますが、私がわかりやすいと思ったのはここ)
イルミナ社の超並列高速シークエンサーによって出される膨大なデータは、ゲノム解析を革命的に進歩させ、ある種、Precision Medicineをリードさせたと思います。
でも、Preciseではあるかもしれないけれど、真の変異をコールできていないかもしれない、というところがネックでした。
スタンフォード大のDr. Deweyらが、JAMAに2014年に発表した論文、「Clinical Interpretation and Implications of Whole-Genome Sequencin, JAMA. 2014;311(10):1035-1045」によると、56の重要疾患遺伝子について、ショートリードで30X読んでも、10本未満のリードしかマップされなかった(読まれなかった)遺伝子は結構多かったらしいです(下図)。
また、想像つくかもしれませんが、ショートリードだけに頼るゲノムリシークエンスでは、
です。
そこで、ロングリードで、低カバレッジで、正確に変異を検出することが大事になってくるわけですね。
先の論文の著者でもある、Dr. Euan Ashleyは、先日Nature Reviews Geneticsにレビューを発表しました。
リンク先はこちらです
パーソナルゲノム、クリニカルゲノムシークエンス、などの分野に携わるひと、これからやろうと思っているひとは、このレビューを読むことをお勧めします!
ちなみにAshley博士は、Personalis社の創始者でもあるのです。Precision Medicine のまさにオピニオンリーダーですね。
またこのレビューの内容の一部は、YouTubeでも公開されているので興味のあるかたはこちらもどうぞ
最後に、Ashley博士は、今年のASHGでも、PacBioのワークショップで講演します!
行くひとは是非チェック!!
これが何を指すのかというと、「現在のゲノム解析技術と知をフルに活用して、個人に合ったオーダーメイド医療、予防医学も含めて、実現しよう!」というものではないでしょうか?
私の理解はそんなところです。皆さんそれぞれ少しずつ違った解釈をされているかもしれませんね。
しかーし、ここで英語の問題を1問。
「PrecisionとAccuracyの意味の違いを述べよ(PreciseとAccurateの違いでも良い)」
さあ、わかりますか?
普通に辞書引くと、どちらも正確性あるいは精度とか書いてあるかもしれません。でも、厳密には違いますよ。
答え:
Precisionとは精度。何回も測定してどれも近い数値が出た場合、精度が高いといい、再現性が高い、とも言う。
Accuracyとは正確度。測定結果が真の値に近い場合、正確度が高いといい、確度が高いとも言う。
なので、いくらPrecisionが良くても(同じゲノムを何回解析してほぼ同じSNVが測定されても)Accurateで無い場合(真の変異では無い)もある
(ググれば例が出てきますが、私がわかりやすいと思ったのはここ)
イルミナ社の超並列高速シークエンサーによって出される膨大なデータは、ゲノム解析を革命的に進歩させ、ある種、Precision Medicineをリードさせたと思います。
でも、Preciseではあるかもしれないけれど、真の変異をコールできていないかもしれない、というところがネックでした。
スタンフォード大のDr. Deweyらが、JAMAに2014年に発表した論文、「Clinical Interpretation and Implications of Whole-Genome Sequencin, JAMA. 2014;311(10):1035-1045」によると、56の重要疾患遺伝子について、ショートリードで30X読んでも、10本未満のリードしかマップされなかった(読まれなかった)遺伝子は結構多かったらしいです(下図)。
 |
| JAMA. 2014;311(10):1035-1045 |
また、想像つくかもしれませんが、ショートリードだけに頼るゲノムリシークエンスでは、
- リードより長いリピート配列は検出できない(3塩基繰り返しリピートなどは検出不可)
- 多型性が高い配列(HLAなど)で数キロに及ぶハプロタイプを求めるのは困難
- 大きな(1000bp以上の)構造変異を検出するのは困難
です。
そこで、ロングリードで、低カバレッジで、正確に変異を検出することが大事になってくるわけですね。
先の論文の著者でもある、Dr. Euan Ashleyは、先日Nature Reviews Geneticsにレビューを発表しました。
リンク先はこちらです
パーソナルゲノム、クリニカルゲノムシークエンス、などの分野に携わるひと、これからやろうと思っているひとは、このレビューを読むことをお勧めします!
ちなみにAshley博士は、Personalis社の創始者でもあるのです。Precision Medicine のまさにオピニオンリーダーですね。
またこのレビューの内容の一部は、YouTubeでも公開されているので興味のあるかたはこちらもどうぞ
最後に、Ashley博士は、今年のASHGでも、PacBioのワークショップで講演します!
行くひとは是非チェック!!
2016年8月16日火曜日
ハイブリッドアセンブリ 悩みどころとそのヒント
リオオリンピック、日本選手の活躍が目立っていますね。
ところで皆さんはどの競技が好きですか? 水泳?マラソン?陸上?
私はやっぱり陸上ですね。
ボルトの100mもすごかったですが、見入ってしまうのは普段あまり目にしない、ハンマー投げとか、円盤投げとか、3000m障害とかですよ。
投げ系は単純に飛距離を競うところがわかりやすいし、投げた後の雄たけびで、こっちも力が入る。
障害はもはや、何でもありでしょう。飛んだり跳ねたり、水辺に入ったり、誰がこんなルール作ったのか。
最初はみんな一緒にスタートするのに、途中で離されたり、転んだり、抜き返したり、ドラマがありますね。
さて、ドラマがあるといえば「ハイブリッドアセンブリ」
使うツールとデータ量のサジ加減で結果がずいぶん違ってくる=ドラマがある
ハイブリッドアセンブリとは、異なる技術のシークエンサーデータを組み合わせてアセンブルすることです。
7年前くらいは、ハイブリッドといえば、数十bpのイルミナリードと、数百bpの454リードを合わせてアセンブルする、ということを良く聞きました、が、
PacBioの登場で、ハイブリッドアセンブリは大きく2つの意味に分かれました
この論文は、PacBioリードのアセンブリ戦略をこれから検討するかたに、いくつかヒントを与えてくれます。
彼らはショウジョウバエのゲノムをサンプルにして、PacBioとイルミナのシークエンスをおのおのどの程度(ゲノムカバレッジ)読んで、どのツールで解析すれば良い結果(ContiguityやCompleteness)を得られるか調べています。
ハイブリッドアセンブリの方は、いろいろ試した結果、DBG2OLCを採用。
Platanusを使って67.4xのイルミナデータ(DPGPプロジェクトデータを拝借)をアセンブリし、そのContigとPacBioロングリード(様々なカバレッジ量)をハイブリッドアセンブリするパイプライン。
比較としてPacBioのみのアセンブリは、PBcRを採用(注:いまはCanuというツールになっています)
ま、詳しくは論文を参照して頂くとして、ネタばれします。(先入観持たずに論文読みたいひとはここから先は読まないで)
---------------------------------------------------------------------------
M&Mの最初に、ショウジョウバエのゲノムを抽出して、サイズセレクションして・・・って書いてあるけど、シークエンスしたデータはこのアセンブル解析には使っていない、ってはっきり書いてある! え?
全ての解析データはpublicly availableのものだって!?
ま、それはみんなが試せるから良いとして、ここはスルー
NG50のグラフ(論文中Fig 5)を見ると、PacBioリード40x、53xまでは、ハイブリッドアセンブリの結果の方がNG50が長い(下のほうの3本はハイブリッド、真ん中の青はPacオンリー、一番上の赤線は両方のマージ)
でもその上、62x、77x、99x、121xのPacBioリードでアセンブリすると、NG50は一気に長くなる!
だからPacデータを使うなら、Pacオンリーデータでアセンブリしたほうが良い!
と、言ってもやっぱりハイブリッドは、需要があるわけです。
理由はコストとサンプル量の制限です。
ほぼ無制限に予算があって、ほぼ無制限にDNAが取れれば、Pacだけでアセンブリしたほうが良いに決まってますが、普通そうはいかない。
この論文でも言っていますが、PacBioを30x程度と、イルミナを70x程度用意できれば、そこそこリーズナブルな結果は得られるでしょう。
この論文の例では、Pacデータ30xと、イルミナデータ70xのハイブリッドの結果のNG50は、Pacオンリーの50xアセンブリのときより長かった、そうです。
もちろんゲノムの複雑さによっては、Pacデータは30xでは足りない、なんてこともあるでしょうが、最初のステップとして、30xはお勧めできます(10xは少ないだろうなあ)。
最後に、必要DNAですが、これは結構盲点かもしれませんね。
マイクログラムオーダーのゲノムDNAを用意して、ようやく数個のSMRT Cellを流せるのですから。必要カバレッジのデータ量を得るために、どれくらいのゲノムDNAを抽出する必要があるのか、これを逆算したら結構な量になるかもしれません。
抽出できるDNA量から、カバレッジの限界を計算して、ハイブリッドを選択するケースもあると思います。
ところで皆さんはどの競技が好きですか? 水泳?マラソン?陸上?
私はやっぱり陸上ですね。
ボルトの100mもすごかったですが、見入ってしまうのは普段あまり目にしない、ハンマー投げとか、円盤投げとか、3000m障害とかですよ。
投げ系は単純に飛距離を競うところがわかりやすいし、投げた後の雄たけびで、こっちも力が入る。
障害はもはや、何でもありでしょう。飛んだり跳ねたり、水辺に入ったり、誰がこんなルール作ったのか。
最初はみんな一緒にスタートするのに、途中で離されたり、転んだり、抜き返したり、ドラマがありますね。
さて、ドラマがあるといえば「ハイブリッドアセンブリ」
使うツールとデータ量のサジ加減で結果がずいぶん違ってくる=ドラマがある
ハイブリッドアセンブリとは、異なる技術のシークエンサーデータを組み合わせてアセンブルすることです。
7年前くらいは、ハイブリッドといえば、数十bpのイルミナリードと、数百bpの454リードを合わせてアセンブルする、ということを良く聞きました、が、
PacBioの登場で、ハイブリッドアセンブリは大きく2つの意味に分かれました
- PacBioロングリードを、イルミナショートリードでエラー補正してからアセンブリする(2012年ごろ登場したPacBioToCA, ECToolsでは主にこの意味)
- イルミナショートリードで作ったContigを、PacBioロングリードでScaffoldする(2013年ごろのPBJellyの登場からはこの意味も加わる)
厳密には、2はアセンブリというよりScaffoldingですが、これもハイブリッドアセンブリと呼ばれることが多いです。
ちなみに研究者のひとと話をするとき、どちらの意味でハイブリッドと呼んでいるのか、最初に確認するようにします。でないと、話がすれ違いになってしまう!
ちなみに研究者のひとと話をするとき、どちらの意味でハイブリッドと呼んでいるのか、最初に確認するようにします。でないと、話がすれ違いになってしまう!
では、最近多い、2の意味でハイブリッドアセンブルするとして、どのデータをどれくらい用意して、どのツールでアセンブルしたら良いのでしょうね?
で、ちょうど良い論文があります!
Chakraborty et al., (2016) Contiguous and accurate de novo assembly of metazoan genomes with modest long read coverage. Nucl. Acids Res.
この論文は、PacBioリードのアセンブリ戦略をこれから検討するかたに、いくつかヒントを与えてくれます。
彼らはショウジョウバエのゲノムをサンプルにして、PacBioとイルミナのシークエンスをおのおのどの程度(ゲノムカバレッジ)読んで、どのツールで解析すれば良い結果(ContiguityやCompleteness)を得られるか調べています。
ハイブリッドアセンブリの方は、いろいろ試した結果、DBG2OLCを採用。
Platanusを使って67.4xのイルミナデータ(DPGPプロジェクトデータを拝借)をアセンブリし、そのContigとPacBioロングリード(様々なカバレッジ量)をハイブリッドアセンブリするパイプライン。
比較としてPacBioのみのアセンブリは、PBcRを採用(注:いまはCanuというツールになっています)
ま、詳しくは論文を参照して頂くとして、ネタばれします。(先入観持たずに論文読みたいひとはここから先は読まないで)
---------------------------------------------------------------------------
M&Mの最初に、ショウジョウバエのゲノムを抽出して、サイズセレクションして・・・って書いてあるけど、シークエンスしたデータはこのアセンブル解析には使っていない、ってはっきり書いてある! え?
全ての解析データはpublicly availableのものだって!?
ま、それはみんなが試せるから良いとして、ここはスルー
NG50のグラフ(論文中Fig 5)を見ると、PacBioリード40x、53xまでは、ハイブリッドアセンブリの結果の方がNG50が長い(下のほうの3本はハイブリッド、真ん中の青はPacオンリー、一番上の赤線は両方のマージ)
でもその上、62x、77x、99x、121xのPacBioリードでアセンブリすると、NG50は一気に長くなる!
 |
| Chakraborty et al. Fig 5 |
と、言ってもやっぱりハイブリッドは、需要があるわけです。
理由はコストとサンプル量の制限です。
ほぼ無制限に予算があって、ほぼ無制限にDNAが取れれば、Pacだけでアセンブリしたほうが良いに決まってますが、普通そうはいかない。
この論文でも言っていますが、PacBioを30x程度と、イルミナを70x程度用意できれば、そこそこリーズナブルな結果は得られるでしょう。
この論文の例では、Pacデータ30xと、イルミナデータ70xのハイブリッドの結果のNG50は、Pacオンリーの50xアセンブリのときより長かった、そうです。
もちろんゲノムの複雑さによっては、Pacデータは30xでは足りない、なんてこともあるでしょうが、最初のステップとして、30xはお勧めできます(10xは少ないだろうなあ)。
最後に、必要DNAですが、これは結構盲点かもしれませんね。
マイクログラムオーダーのゲノムDNAを用意して、ようやく数個のSMRT Cellを流せるのですから。必要カバレッジのデータ量を得るために、どれくらいのゲノムDNAを抽出する必要があるのか、これを逆算したら結構な量になるかもしれません。
抽出できるDNA量から、カバレッジの限界を計算して、ハイブリッドを選択するケースもあると思います。
2016年7月12日火曜日
バクテリアのIsoSeqをするには?
今日はバクテリアのIso-Seqの話です。
そもそもイントロンが無いバクテリアの転写産物ですが、完全長で読む意義はあります、よね。完全長で読みたい、というニーズはあったようです。
ではどうするか?
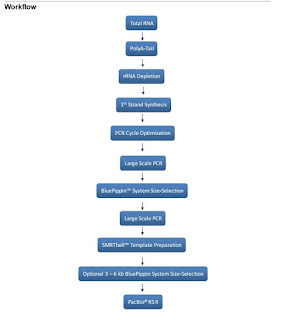
CDNAを作る逆転写には、タカラクローンテック社の SMARTer PCR cDNA Synthesis kit を使います。
でもこの逆転写反応には、PolyA-Tail 配列があることが必要です。
しかしバクテリアのmRNAには、PolyA-tail 配列が無い!
そこで最初に、mRNAの3'側に、酵素反応で強制的にPolyA-tail 配列をつけてしまいます。
Epicentre PolyA Polymerase Tailing Kit
しかしこの反応は、rRNAに対しても PolyA-Tail を付加してしまうので、この後 rRNAを除去することが必要です。
Thermo Fisher RiboMinus
流れとしてはこんな感じ
二重鎖 cDNA が得られたら、PCR増幅して、サイズセレクションして、SMRT Bell ライブラリ作って、RSIIでシークエンス
SMRT bell作る前処理の、rRNA probe とのハイブリ効率はどれくらいなんでしょう?
ここでちゃんとrRNAを除いておかないといけませんね。
プロトコルは、Unsupported つまり、サポート対象外ですが、興味のあるかたはここからご覧下さい
2016年7月11日月曜日
CCSでアセンブリも、あり!
毎日暑いですね。
そんな時はアイスかかき氷!
私は普段、冷たいものはあまり食べないですが、こうも毎日暑いと食べたくなりますよ。
「黒糖ぜんざい」
沖縄で食べたかきごおりです。ぜんざい、と呼ぶそうですが。
これはおいしかった。
さて、PacBioでのゲノムアセンブリといえば、ロングリード(CLR)を互いにエラーコレクションして行なうのが「定説」と思っている方も多いと思います。
CCSというのは、Circular Consensus Sequenceの略で、比較的短いライブラリを何度も読んで、1分子のDNAの精度を高めていく方法。
何回も同じDNAを読むことで、出力された配列のコンセンサス配列(1分子DNA由来)を、99%以上に高めることができるのです。
今の試薬P6-C4なら、5パス以上で99%の精度に達することも可能です。
ライブラリの長さにもよりますが、1kbから2kbなら、十分精度の高いリード(CCS)を出力することも可能で、それを使ってアセンブリを行なった論文がこちら
8個のSMRT Cell から得られた平均 1,319 bp、99.7%の精度(平均10パス)、94 Mb分のCCSを使って、アセンブリ。
アセンブラーは、MIRA 4.0
この論文はメタゲノムなので、完全ゲノム配列を作るのが目的ではありません。
マーカー遺伝子が読めていればOK
Phylotype Specific な配列データベースを作成するには、どんな配列もバイアス無く読めるPacBioが最適というわけです。
CCSを使った、アセンブリの論文でした。
そんな時はアイスかかき氷!
私は普段、冷たいものはあまり食べないですが、こうも毎日暑いと食べたくなりますよ。
「黒糖ぜんざい」
沖縄で食べたかきごおりです。ぜんざい、と呼ぶそうですが。
これはおいしかった。
さて、PacBioでのゲノムアセンブリといえば、ロングリード(CLR)を互いにエラーコレクションして行なうのが「定説」と思っている方も多いと思います。
CCSというのは、Circular Consensus Sequenceの略で、比較的短いライブラリを何度も読んで、1分子のDNAの精度を高めていく方法。
何回も同じDNAを読むことで、出力された配列のコンセンサス配列(1分子DNA由来)を、99%以上に高めることができるのです。
今の試薬P6-C4なら、5パス以上で99%の精度に達することも可能です。
ライブラリの長さにもよりますが、1kbから2kbなら、十分精度の高いリード(CCS)を出力することも可能で、それを使ってアセンブリを行なった論文がこちら
8個のSMRT Cell から得られた平均 1,319 bp、99.7%の精度(平均10パス)、94 Mb分のCCSを使って、アセンブリ。
アセンブラーは、MIRA 4.0
この論文はメタゲノムなので、完全ゲノム配列を作るのが目的ではありません。
マーカー遺伝子が読めていればOK
Phylotype Specific な配列データベースを作成するには、どんな配列もバイアス無く読めるPacBioが最適というわけです。
CCSを使った、アセンブリの論文でした。
2016年7月6日水曜日
パイナップルドラフトゲノム PacBioでアセンブリ
皆さん、パイナップルって、よく食べます?(私はたまーにしか食べません)
日本では、リンゴやブドウほどメジャーではないせいか、パイナップルの品種名って、あまり知らないですよね。
八百屋やスーパーでも、普通に「パイナップル」だったりするし。
恥ずかしながら、今日まで、パイナップルにたくさんの種類があるなんて気にしたこともありませんでした。
サイトはこちら
世界で一番多く栽培されているのが、Smooth Cayenne(スムース・カイエン)種だそうです。
でも今、MD-2、別名ゴールデン・パイナップル、スーパー・スイート、などと呼ばれている品種が次々と栽培されているらしい。
時は1961年、ドール社、デルモンテ社、マウイ・パイナップル・カンパニーの出資によって、ハワイ州に、Pineapple Research Institute(PRI)が設立されました。
そこで品種改良によって生まれたのが、このMD-2品種。MDの名は、パイナップル・カンパニーのゼネラルマネージャーの妻の名前、Millie Dillardさんに由来するそうです。
(以上、UTAR AGRICULTURE SCIENCE JOURNAL ● VOL. 1 NO. 4. OCTOBER 2015からの情報でした)
パイナップルって、今まで考えたことなかったのですが、受粉しなくても果実を作るそうですね。
Parthenocarpy(単為結果)と呼ぶそうです。バナナと同じ。
そして、non-climacteric な植物。
これは、果実を収穫してからエチレンガスをほとんどあるいは全く出さないので、収穫後に果実が熟すことはないらしい。ここのサイトが詳しい。
そういえば、収穫後にエチレンガスを噴霧して、食べ頃をコントロールする話は、昔聞いたことがありました
そんなパイナップルですが、MD-2のドラフトゲノムが論文になりました。
Biotechnology Research Institute, University Malaysia Sabah の、Dr.Redwan らの仕事です
PacBioを使ったアセンブリプロジェクトです。
ここではイルミナリードも使われていますが、目的はPacリードのエラー補正と、Pacアセンブリ後のContigの、Scaffoldingのみです。
先ず、イルミナライブラリ用に350bpと550bpのインサートを作り、HiSeqを使って100bpのシングルまたはペアエンドシークエンス。
さらに、750bpのインサートをMiSeqを使って300bpのヘアエンドシークエンス。
Q20のクオリティ、最低50bpのリードを解析に使用して、154.7カバレッジのデータ量を得た。
PacBioは20kbライブラリ、P4-C2、P5-C3で32セルシークエンス。
フィルタリング後の11.78Gbデータを、イルミナの350pb、750bpライブラリデータで補正。
これには、novoLR package(Novocraft ソフトウェア)を使用。
補正後のデータは8.34Gb(15.9 x)になったので、これをCelera Assemblerにかけてアセンブリ → N50 = 25,277bp
Contigの冗長性を除くため、25,000bp以下の短いContigをそれ以上の長いContigにマップして80%以上のContigを除いたり、エラー補正後のリードをContig配列にマップしたりして16%冗長配列を除いている。
このような努力の結果、トータルのContigサイズは想定ゲノムサイズの 96.6% の 508 Mb、N50 は34,762 bpになった。
このようにして作られたContigは、次にイルミナリード、補正前のPacBioリードを利用して、novoLRpolishというソフトでScaffoldingし、N50 が153,084bp、最大Scaffoldが1.29Mbになり、トータル塩基数は524Mbpになった。
アセンブル後のゲノム配列の評価には、CEGMA(Core Eukaryotic Genes Mapping Approach)というツールを使っている。
簡単に言うと、真核生物に存在するコアな遺伝子たちがちゃんと読めているか、を評価するデータツールです。
しかし、残念ながら、CEGMAは昨年サービスを中止したらしいです。
これからはBUSCO(Benchmarking Universal Single-Copy Orthologs)らしいです。
さてさて、もうひとつ今日驚いたこと。
パイナップルの果肉は、果実じゃない!
ちなみにパイナップルを使った料理のレシピは、クックパッドによれば、現在6,000種類以上もあります!!
こちらも驚き!
登録:
投稿 (Atom)